【科研小助手】RnaSeqSampleSize:RNA-Seq样本量评估工具
随着高通量测序技术的发展,RNA-Seq已经成为差异基因表达研究中的常规检测方法。RNA-Seq实验设计最重要内容之一就是选择最佳生物重复数以获得所需统计效能(sample size estimation,样本大小估计),或者估计在数据集中成功发现统计意义的可能性(power estimation,效能估计)。重复次数不足可能导致结论不可靠,而重复次数过多可能导致时间和资源浪费,因此需要研究者在研究成本和实验效能之间寻求权衡。
几种常用估计方法及不足
因为RNA-Seq数据可用read counts表示,所以在早期的RNA-Seq研究中,分析主要基于泊松分布(Poisson distribution)进行。然而泊松分布不能很好的与经验数据相吻合,这主要是由于生物自然变异的过度离散引起的。为了解决这个问题,基于负二项分布的方法(negative binomial distribution-based methods)被开发出来,为样本间的变异配置了更大的灵活性。后来,人们又陆续基于单基因差异表达、多个基因的比较,以及将预算成本纳入分析方法,开发出RNASeqPower、PROPER等多种评估工具。
然而,以往方法仍然存在一些局限性,例如:没有正确考虑平均read counts和不同基因的离散程度,缺乏适当的参考数据,以及缺乏简单和用户友好的界面。因为基因的平均read counts分布在四个数量级以上,它们的离散高度依赖于它们的基因表达水平。由于以前的估计方法不是为这样的分布设计的,所以研究者经常使用保守地或者根据经验选择的一个值,这经常导致样本量估计过高。虽然近期有研究考虑了基因表达水平与其离散性之间的相关性,引入了一种基于模拟的程序,但这种方法尚未开发出易于使用的软件工具。并且,这些方法数据量巨大,占用大量计算资源,也不适用所有规模的项目。
RnaSeqSampleSize优势:
由于上述问题的存在,该工具开发者基于多重检验中的FDR错误控制,并利用真实数据的平均read counts和离散分布来估计更可靠的样本大小。
开发出R语言工具包及在线工具RnaSeqSampleSize,在线网址为:
https://cqs.mc.vanderbilt.edu/shiny/RnaSeqSampleSize/。
网站首页展示:
相关研究发表在2018年5月30日的BMC Bioinformatics上。
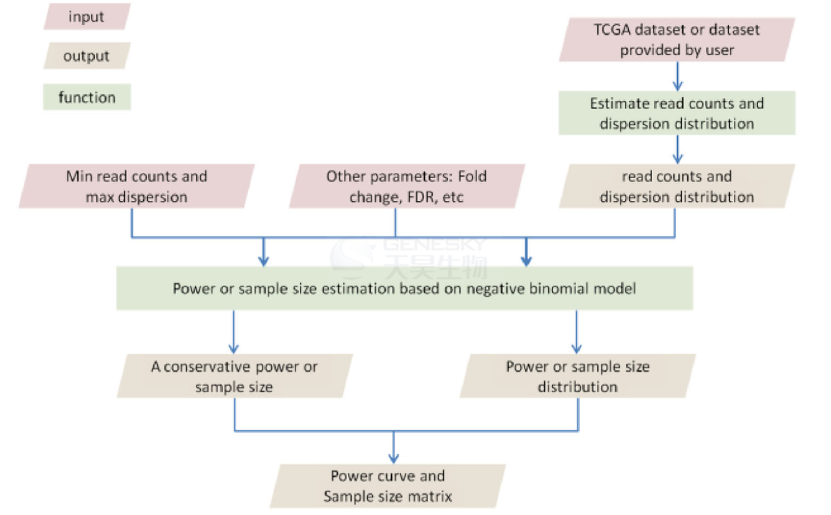
图1、RnaSeqSampleSize工作流程
RnaSeqSampleSize首先利用如癌症基因组图谱(TCGA)数据库中真实的RNA-Seq数据集来估计基因平均read counts和离散分布。RnaSeqSampleSize可以利用大规模平均read counts和离散程度数据估计,支持多达2000个平均read counts,利用这些信息来指导样本量和功效的估计。此外,RnaSeqSampleSize还具有几个独特的特点,包括对感兴趣的基因或途径的估计、功效曲线可视化和参数优化等。
基于真实数据的样本量估计
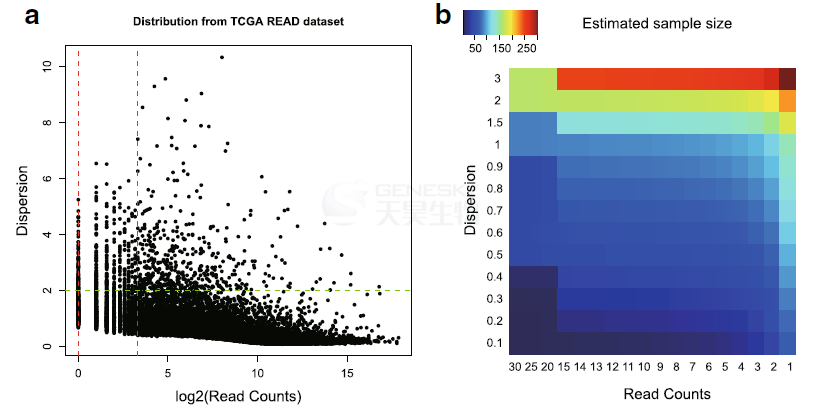
如前所述,基因的平均read counts和离散程度在单个RNA-Seq实验中具有广泛的分布。平均read counts或离散的微小波动将极大地影响估计的功率或样本大小(图2)。例如,在TCGA直肠腺癌(READ)数据集中,基因具有从0到10的离散度,并且平均read counts从1到数千 (图2a)。在这种情况下,从单个值估计样本大小是不准确的。本研究计算出,当最小平均read counts从1变为30,最大离散从0.1变为3时,估计的样本大小从10增加到302 (图2b)。
图2、Read counts或离散对估计的样本大小和功效有很大影响。a. TCGA直肠腺癌(READ)数据集中所有基因的read counts和离散分布。红线表示read counts等于1和10。绿色线条表示所有基因离散的95%。b. 在read counts或离散的不同组合中实现0.8的power值所需的估计样本大小
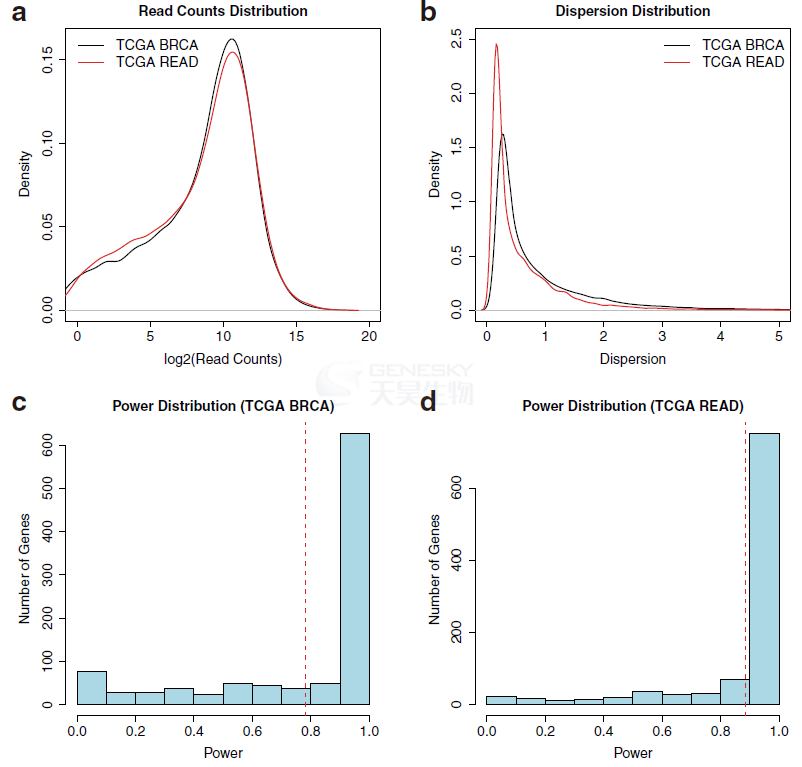
图3、用真实数据估计样本大小。a. TCGA乳腺浸润性癌(BRCA)和直肠腺癌(READ)数据集中所有基因的read counts分布;b. TCGA BRCA和READ数据集所有基因的离散分布;c. 当样本大小等于71时,TCGA BRCA数据集中基于计数和离散分布的功效分布。红线表示power平均值。d .当样本大小等于71时,基于TCGA READ数据集中的read counts和离散分布的功效分布。红线表示power平均值
感兴趣基因或途径的样本量估计
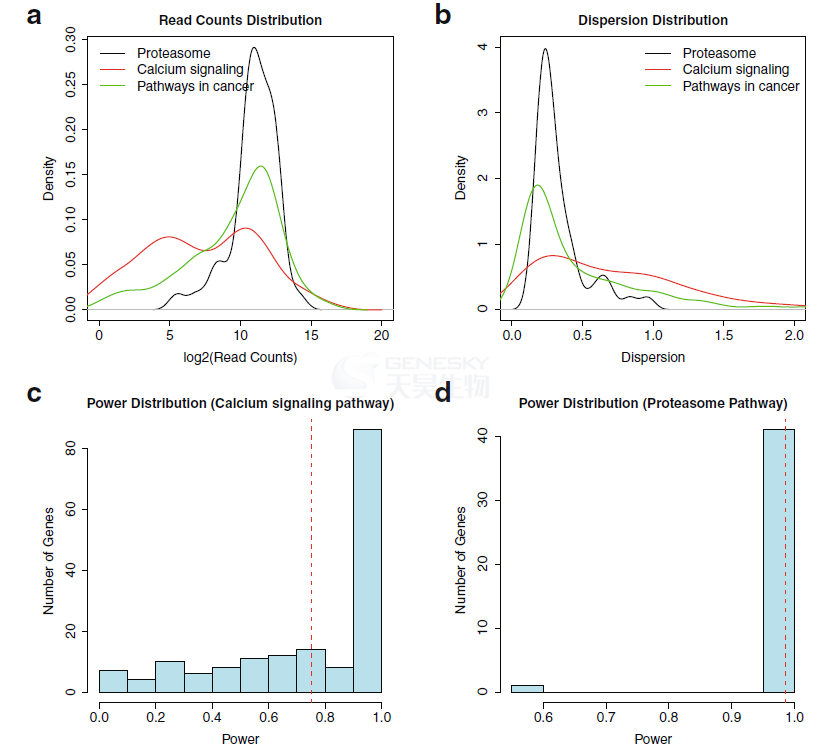
在某些情况下,研究人员可能对某些特征(如共享通路或基因GO类别)定义的基因子集感兴趣,而不是对整个基因组感兴趣。在这种情况下,样本量估计方法需要调整,因为与其他基因相比,感兴趣的基因子集可能具有不同的表达模式。RnaSeqSampleSize被设计成通过允许用户提供感兴趣基因的列表或KEGG通路ID来处理这样的实验设计中的样本大小和功效分析;这确保了只有感兴趣的基因或所选途径中的基因的read counts和离散分布被用于估计(图4)。
图4、感兴趣基因的样本量估计。a. TCGA READ数据集中三个KEGG通路基因的的read counts分布;b. TCGA READ数据集中三个KEGG通路中基因的离散分布; c. 当样本大小等于71时,基于TCGA READ数据集中钙信号通路基因的计数和离散分布的功效分布。红线表示power平均值。d.当样本大小等于71时,基于TCGA READ数据集中蛋白酶体途径基因的计数和离散分布的功效分布。红线表示power的平均值
不同参数下的功效曲线可视化及优化
功效曲线被广泛用于分析和比较样本大小估计结果。为了演示RnaSeqSampleSize中的功率曲线可视化特性,研究者根据不同的场景生成了三条功率曲线。如图5a所示,X轴表示两组中使用的样本总数,Y轴表示估计功效。样本分配设计有三种类型:两组1:1样本大小(红色曲线);2:1两组样品大小(蓝色曲线);3:1两组样品大小(紫色曲线)。功效和样本数之间的关系可以很容易地可视化。在图5a所示的例子中,功效曲线表示当使用相同的样本总数时,平衡(样本大小1:1 )实验设计(红色曲线)获得最高功效。
图5、用RnaSeqSampleSize实现功效曲线可视化和参数优化。平衡后(两组样本大小相同)和未平衡(两组样本大小不同)的实验设计功效曲线。功效曲线表明,平衡后实验设计(红线)在相同样品总数下将获得最高功率;b. 样本量估计中的参数优化。离散和倍数变化分别设置为0.5和2。生成具有不同样本数和读取计数对的功效矩阵。功效分布表明,样本数对功效的确定起着更重要的作用,建议在RNA-Seq实验中至少使用96个样本,利用这些参数得到0.8的功率
RNA-Seq实验设计经常受到预算的限制。RnaSeqSampleSize中的优化功能可用于确定在不超出预算的情况下实现最高功率的最佳参数。为了演示参数优化功能,研究者尝试优化样本数和read counts,同时固定所有其他参数(fold change: 2;离散度: 1;FDR : 0.05 )通过产生功率矩阵(图5)。当使用16个样本时,即使读取计数高达96,估计功率也小于0.1。然而,当样本数增加到96时,即使当读取计数低至8时,估计功率也增加到0.8。该矩阵表明,样本数量在确定power方面比read counts起更重要的作用。
往期精选文章:
37个RNA-seq工具大PK,教你数据处理方法如何选择
【昊阅读】基因表达数据的模块检测方法综合评价
TCGA收官之作—27篇重磅文献绘制“泛癌图谱”
【昊阅读】RNA-seq揭示油茶冷适应的分子机制
LncRNA研究,像这个夏天一样“火热异常”
关于天昊:
天昊生物,RNA-seq技术的优质服务提供商!我们通过对RNA-seq各个实验及生信分析环节不断优化,为客户提供更加准确、可靠及个性化的数据检测和分析结果,为您的科学研究保驾护航!

 咨询热线:400-065-6886
咨询热线:400-065-6886

 咨询热线:400-065-6886
咨询热线:400-065-6886