癌症研究方案
Part Ⅰ:癌症研究思路
癌症又称为恶性肿瘤,作为复杂疾病的典型代表,其发生是在多基因、多因素、多阶段共同作用造成的细胞功能紊乱,异常增殖形成的新生物(Neogrowth)。
由于癌症的特点是整个信号网络发生改变,如多个基因发生突变、多条信号通路同时发生改变,因此癌症研究就要求检测错综复杂的大量基因突变/调控改变(高通量),同时必须能够精准的检测出低百分比的改变(高精度)。二代测序已经广泛应用于癌症研究,正是由于其具备的高通量、高灵敏度的特点,尤其是一次实验中挖掘出多个有意义的研究内容。
癌症的研究,主要集中在诊断、治疗效果、预后、复发转移四个领域。无论开展癌症患者与正常人直接的差异研究,还是针对同种癌症患者间诊断、疗效、预后、复发转移的差异,只是在病人选取时有差异,研究方法并没有太大差别。
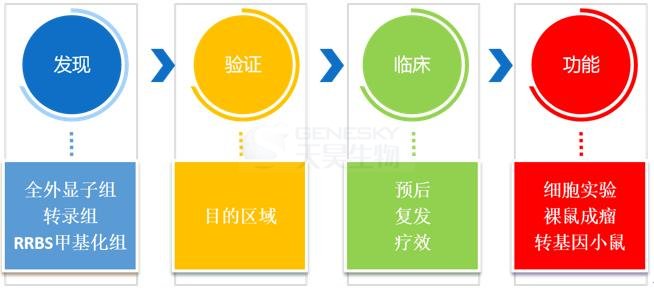
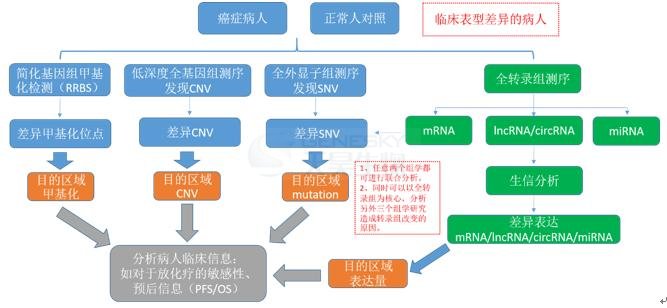
癌症研究的基本流程如下:
1.在初始的发现阶段,使用二代测序技术可以一次筛选出整个组学层面的改变。
2.在验证阶段,对于选定的差异基因,进行高深度的目的区域测序。
3.无论上述组学或目的区域测序,拿到的结果,是为了解释临床问题,所以一定要回归临床表型,进行临床信息的相关性分析。
4.通过上述测序并进行临床分析后得到的关键基因,可以进行成熟套路的体外培养细胞或小鼠实验,阐明其调控机理,进一步提升文章的影响力。
Part Ⅱ:组学研究
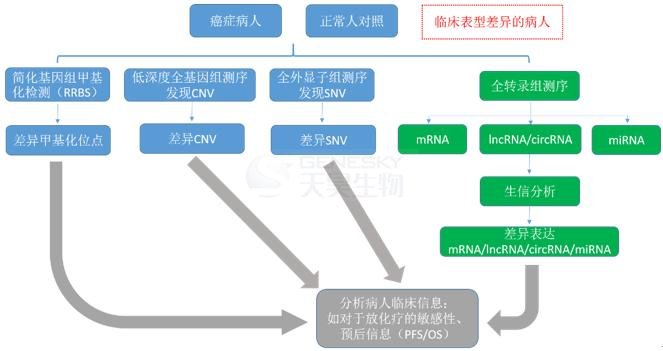
在癌症研究的发现阶段,使用二代测序技术可以一次筛选出整个组学层面的变化:
1、全基因组测序(WGS)或全外显子组测序(WES)发现基因组的SNP(single nucleotide polymorphism)、mutation、CNV(copy number variation)等差异;
2、转录组测序发现在癌症发生发展过程中发挥重要作用的差异mRNA、miRNA、lncRNA、circRNA;
3、全基因组甲基化测序(WGBS)或简化基因组甲基化测序(RRBS),发现癌症患者发生的表观遗传学改变。
二代测序可开展的内容
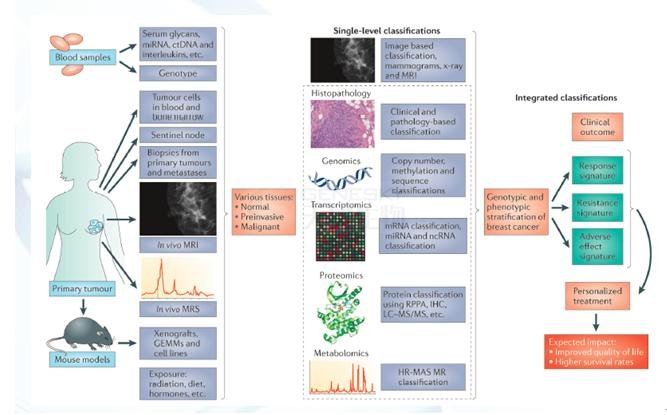
目前研究火热的癌症分子分型、多组学研究,就是将上述多个组学层面的研究结合在一起,全面分析癌症发生发展过程中DNA和RNA发生的改变。如:胃癌转录组结合CNV和甲基化进行分子分型,最后发现其中一组病人使用5-FU进行化疗反而预后更差。
多组学研究思路
Part Ⅲ:目的区域测序
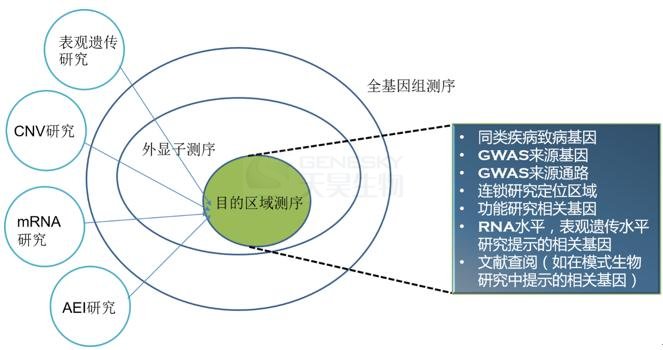
由于组学测序的深度较浅(如全基因组测序深度一般为30x左右、全外显子测序一般为200x左右),对于选定的差异基因,进行高深度的目的区域测序,能找到与癌症发生发展过程密切相关的低频基因改变(SNP、mutation、CNV等)或甲基化改变。
目的区域测序除了能提供测序深度更高、更准确的测序结果以外,另一个重要特点就是与整个组学测序相比,结果更好解读,因为结果只包含预先选定的基因。
并且目的区域测序具有更好的应用价值,如目前火热的液体活检,也是采用目的区域测序方法,对癌症病人进行预先选定基因的高深度测序,进而发挥帮助病人选择靶向药等作用,在临床诊治过程中发挥重要作用。
最为重要的是,与组学检测相比,目的区域无可比拟的优势就是,便宜!几乎每种癌症都有若干篇大牛发表的多组学或单一组学文章,对于我们而言,同样的花费下,检测几个样品的组学层面改变,或许并没有从文献中选取基因并进行高深度目的区域测序效果更好。
天昊生物客户,采用全基因组测序加高深度目的区域测序,发表了Nature Communication; 另一位天昊生物客户,通过对预先选定的甲基化位点进行目的区域测序,一步简单实验同样发表Clinical Epigenetics.
目的区域可开展的内容
目的区域研究思路
使用目的区域测序,癌症研究多、快、好、省!
超过50,000份样品的目的区域测序经验,信任源自专业!
Reference:
• Principles and methods of integrative genomic analyses in cancer. Nat Rev Cancer. 2014 May; 14(5):299-313.
• Identification of Molecular Subtypes of Gastric Cancer With Different Responses to PI3-Kinase Inhibitors and 5-Fluorouracil. GASTROENTEROLOGY 2013; 145: 554–565.
天昊生物复杂疾病研究成功案例:
1. Whole-exome and targeted sequencing identify ROBO1 and ROBO2 mutations as progression-related drivers in myelodysplastic syndromes.
Nature communication. 2015, 26; 6: 8806.
2. Identification and validation of the methylation biomarkers of non-small lung cancer(NSCLC).
Clinical Epigenetics (2015) 7:3.

 咨询热线:400-065-6886
咨询热线:400-065-6886

 咨询热线:400-065-6886
咨询热线:400-065-6886