SSR在基因精细定位中的应用
摘要: SSR与精细定位
SSR (Simple Sequence Repeats,简单序列重复)是一种常用的遗传分子标记。之前小编给大家介绍了SSR在遗传多样性、群体结构和性状关联分析中的应用(
学会工具“三件套”,SSR分析不再难)。除此之外,SSR还可以在基因的精细定位中发挥作用。
要做到“精细定位”,既需要该区间存在足够多的分子标记,又需要有足够多的重组事件发生,二者缺一不可。没有足够的分子标记,就无法进一步缩小区域范围;没有重组事件发生,则无法衡量分子标记与目标基因的距离。要获得高密度的标记,通常需要在基因组或者转录组测序获得序列信息后,利用相应生信工具进行标记开发;要增加重组事件发生,往往要靠扩大群体来实现。下面小编就以大豆花叶病毒(SMV)抗性基因精细定位为例,跟大家分享两篇近期发表案例。
1、大豆花叶病毒新的抗性基因Rsc15的精细定位及鉴定
发表时间:2017年8月 发表期刊:Theor Appl Genet. 影响因子:4.132
研究背景:
大豆花叶病毒是一种严重危害大豆产量的病害,前期研究发现有些大豆品种具有很强的抗病表型,后发现多个抗病基因(
Rsv1,Rsv3, Rsv4,Rsc3,Rsc4,Rsc7,Rsc8, Rsc14)。作者前期发现一个新的基因
Rsc15,本文在此基础上利用构建的RIL群体对其进行精细定位。
样品选取:
抗病品种RN-9与易感品种7605杂交后的F1、F2 (315个单株)及RIL群体(216 个F
7:11个株系植株)。
SSR标记的选择:
15个SSR标记用于初定位结果的验证 (Satt640, Satt227,Sat_153, Satt322, Sat_213, Sat_246, Satt286, Satt277,Satt557, Satt658, Satt100, Satt708, Sat_238, Staga001, Satt433),132 SSR标记用于精细定位(108个区域SSR标记+用SSRHunter软件新开发的24个SSR标记)。
表型鉴定:
表1、不同群体材料抗病性表型鉴定
表1结果表明,F2群体抗病和不抗病植株为3:1,RILs群体抗病和不抗病植株为1:1,经过卡方检验,与孟德尔分离比率一致,表明该性状主要有一个主效基因控制。
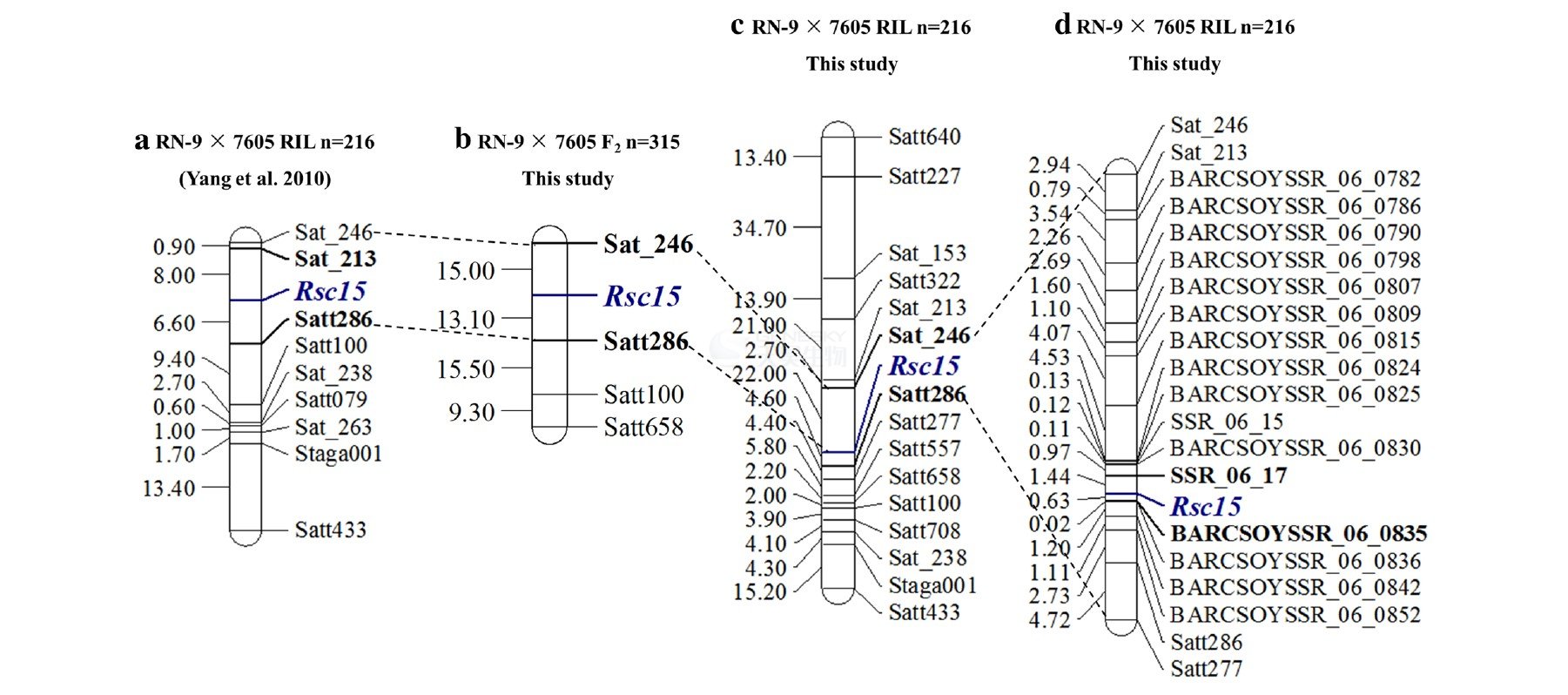
利用RIL群体进行精细定位:
图1、遗传连锁图谱
前期研究,将
Rsc15初步定位在6号染色体Sat_213和Satt286之间的14.6 cM (~1.6 Mb) 区域(图1)。通过在父母本植株中对132个SSR多态性检测,发现18个SSR具有多态性,用于216个RIL群体株系基因型检测,最终利用Join Map 4.0及MapChart 2.2软件构建遗传图谱,根据最小对数似然比LOD为3建立连锁群,最终把
Rsc15定位在了SSR_06_17和 BARCSOYSSR_06_0835之间(图1)。
图2、SMV抗性基因(Rsc15)的遗传和物理图谱
通过分析标记在大豆Williams 82参考序列 (Glyma Wm82.a2.v1)中的位置,确定了
Rsc15位于95Kb的一段区域内(图2-c),包含5个预测基因,通过测序发现只有
GmPEX14 (
Glyma.06g182600)具有多态性,除此之外,侧翼区域的
Glyma.06g175100, Glyma.06g183500 和
Glyma.06g184400也具有多态性,也归为潜在的候选基因(图2-d, 表2)。
表2、候选基因列表
后面通过在不同组织及不同病毒处理时间下候选基因表达检测,以及对抗逆相关的H
2O
2浓度、CAT和POD活性检测,最终发现它们之间存在着高度的相关性(图3)。
图3、RN-9中GmPEX14表达水平与H2O2浓度、CAT和POD活性的相关性分析
文章总结:
整篇文章从新SSR分子标记的开发、扩大的RIL群体构建,到从抗病表型和基因型检测,再到精细定位后候选基因表达及后续功能分析一气呵成,还是比较完整的一篇文章。美中不足就是在功能方面,如果能将抗病品种RN-9中的
GmPEX14转到易感品种7605中,并分析其抗病能力的话,文章将更上一个台阶。
2、具有SMV抗性的大豆品种Suweon97相关基因Rsv1-h的精细定位
发表时间:2016年8月 发表期刊:Theor Appl Genet. 影响因子:4.132
样品选取:
对SC6-N 和SC7-N具有抗性的抗病品种Suweon 97与易感品种Williams 82,及其杂交后的F1、F2 (267个温室种植单株及1150个大田种植单株)、F
2:3(73个单株)和F
3:4(13个株系)群体。
图4、精细定位流程图
SSR标记的选择:
在13号染色体的之前定位的
Rsv1区域发现有128个SSR标记,22个在父母本中显示出多态性。
表型鉴定:
表3、F2群体抗病性表型鉴定
表3结果表明,F2群体抗病和不抗病植株为3:1经过卡方检验,与孟德尔分离比率一致,表明该性状主要有一个主效基因控制。
为了验证是否真正连锁,选用13_1103和13_1187两个标记对1,150 F2单株进行群体基因型检测,在Suweon 97和Williams 82中具有相同基因型的分别记为1103
SS1187
SS和1103
WW1187
WW,最终分别得到262株和285株。自交后得到F3代,随机选取100粒种子种植后进行抗病表型检测,最终验证了之前的连锁结果(图4)。
图5、重组F2代单株基因型物理图谱及对应F2:3植株抗病表型
此外,还检测到529株F2
1103SW1187SW杂合植株,另外74株在13_1103和13_1187两个标记之间发生重组(1103
SS1187
WW或1103
WW1187
SS),然后利用剩余20个SSR对这74个重组植株进行基因型检测,进一步寻找重组交叉点,最终将
Rsv1-
h定位在Suweon 97品种的13_1114和13_1115附近区域(图5)。
图6、13个重组F3:4株系基因型物理图谱及对应抗病表型
种植13个F
2:3株系,选取在重组位点左右两段均为纯和且分别来自父母本的F
3:4进行表型鉴定,进一步验证了
Rsv1-
h在Suweon 97品种的13_1114和13_1115附近区域(图6)。
表4、候选基因列表
最后,本研究根据定位到的~97.5Kb区域进行基因组分析,寻找到8个可能的候选基因,其中2个可能与抗病有关。文章并未对基因的功能进行深入探讨。
通过对以上两篇文章的解析可以发现,利用SSR分子标记进行基因精细定位的思路十分清晰:构建群体,筛选有多态性的SSR,基因型及表型分析,遗传及物理图谱定位,根据基因组信息列出候选基因,进行表达量等功能分析。思路流程理清了马上开始着手实验,一篇4分多的文章就在不远的前方!
往期精选文章:
想测SSR,想到创新技术—SSRseq
SSR分型四大天王,哪个才是适合多倍体的那个它?
SSR动植物中从事的三大行业
SSR究竟能干些啥?
聊聊SSR•遗传多样性
聊聊SSR•遗传多样性(二)
关于天昊:
您还在为“精细定位”而悲催的灌胶跑胶、数条带和比大小吗?赶快从“效率低下的重复性工作中解脱出来吧”!天昊生物自主研发的基于二代高通量测序平台的SSRseq技术,具有更高通量和更高准确性的技术优势,非常适合对10个以上SSR位点、100个以上样本的检测。不仅仅能够获得“片段大小”,更可以把样本的“序列信息”和“等位基因型的比例信息”一股脑儿的提供给您,是SSR检测的“终极”解决方案。我们具有丰富的项目经验,期待成为您SSR基因分型的优质服务提供商!

 咨询热线:400-065-6886
咨询热线:400-065-6886

 咨询热线:400-065-6886
咨询热线:400-065-6886