
 咨询热线:400-065-6886
咨询热线:400-065-6886

 咨询热线:400-065-6886
咨询热线:400-065-6886

In [1]:
install.packages('docopt')
In [2]:
install.packages('plotly')
In [3]:
library(docopt)
library(plotly)

读取pca数据
In[4]:
pca_data = read.table('sample_pca.txt', header = T, check.names = F, stringsAsFactors=F)
head(pca_data)
Out[4]:
sample pc1 pc2
1 0.24040200 0.07686390
10 -0.00802218 0.01788500
100 -0.05724530 0.12847300
101 -0.14421900 -0.13638600
102 -0.06634530 0.00129863
103 -0.02240210 0.09180990
读取分组信息
In[5]:
group_data = read.table('group.xls',sep = ' ' , header = T, check.names = F, stringsAsFactors=F)
head(group_data)
Out[5]:
sample group sub_group
1 A A1
2 A A1
3 A A1
4 A A1
5 A A1
6 A A1
合并pca数据和分组信息
In[6]:
data_plot = merge(pca_data,group_data,by = 'sample' )
head(data_plot)
Out[6]:
sample pc1 pc2 group sub_group
1 0.24040200 0.07686390 A A1
2 0.02387160 0.01739140 A A1
3 0.00757584 0.12740900 A A1
4 0.11741900 0.00890982 A A1
5 0.19129100 0.01753790 A A1
6 0.03781980 0.17492200 A A1

颜色板设置
In[7]:
color_map = c('darkmagenta', 'red', 'green', 'yellow', 'cyan', 'blue')
点的大小和透明度设置
In[8]:
size=1alpha=1
3.1 按照group添加颜色
In[9]:
p1 = plot_ly(data_plot, type = 'scatter', x = ~pc1, y = ~pc2, color =~group,
text =~sample, size = size, alpha = alpha, colors =color_map)
In[10]:
htmlwidgets::saveWidget(p1, 'output1.html', selfcontained = T)
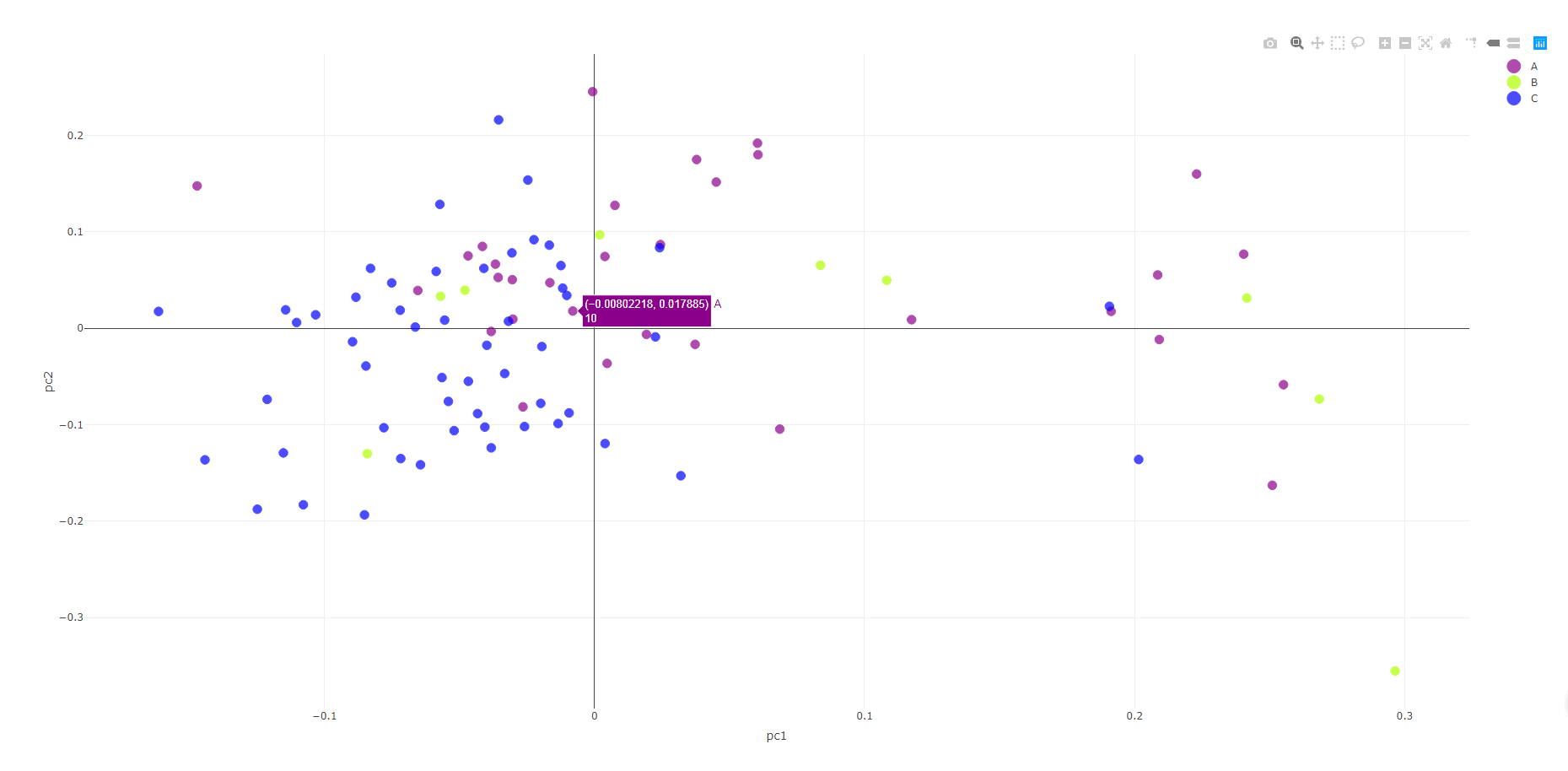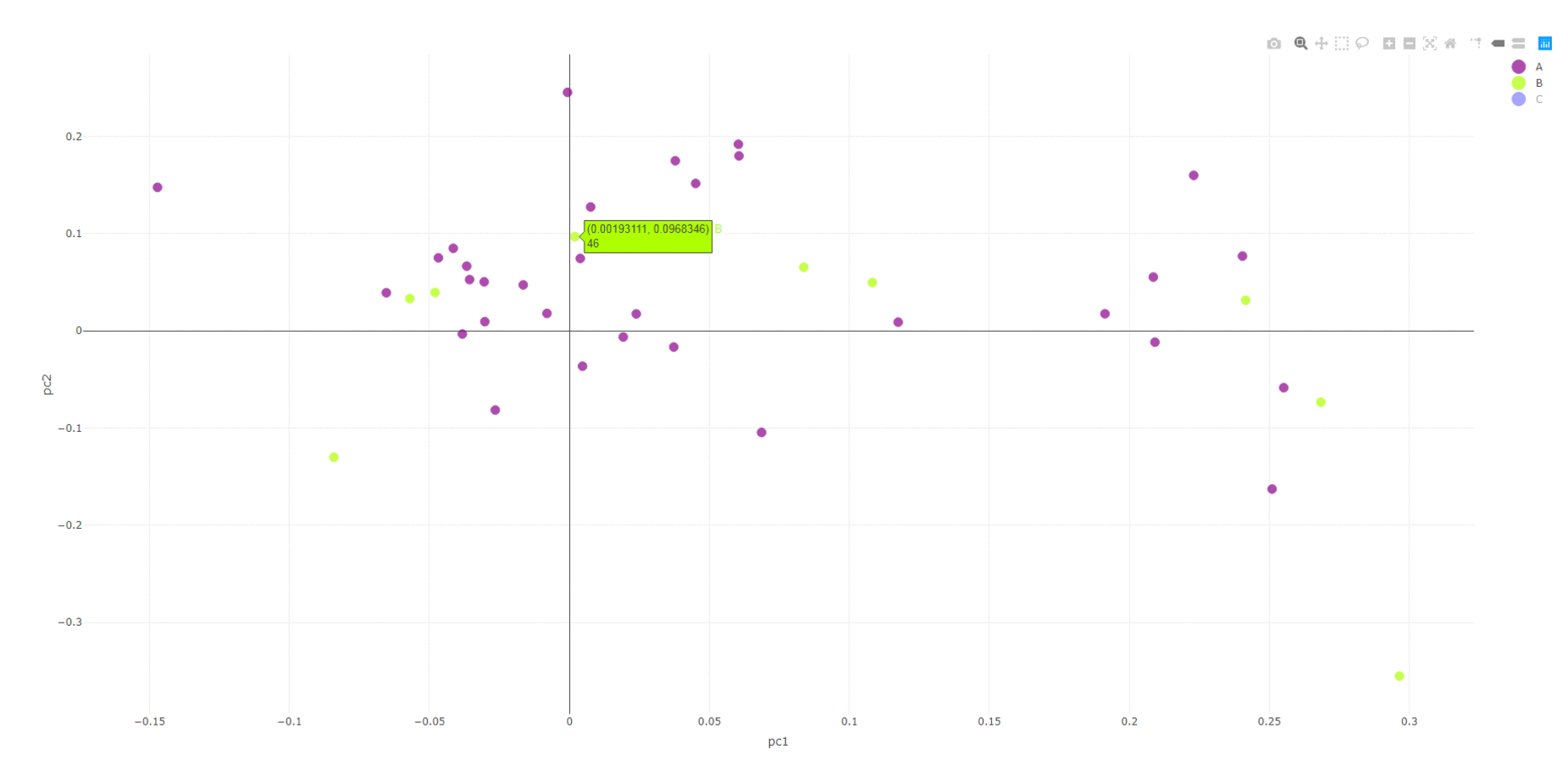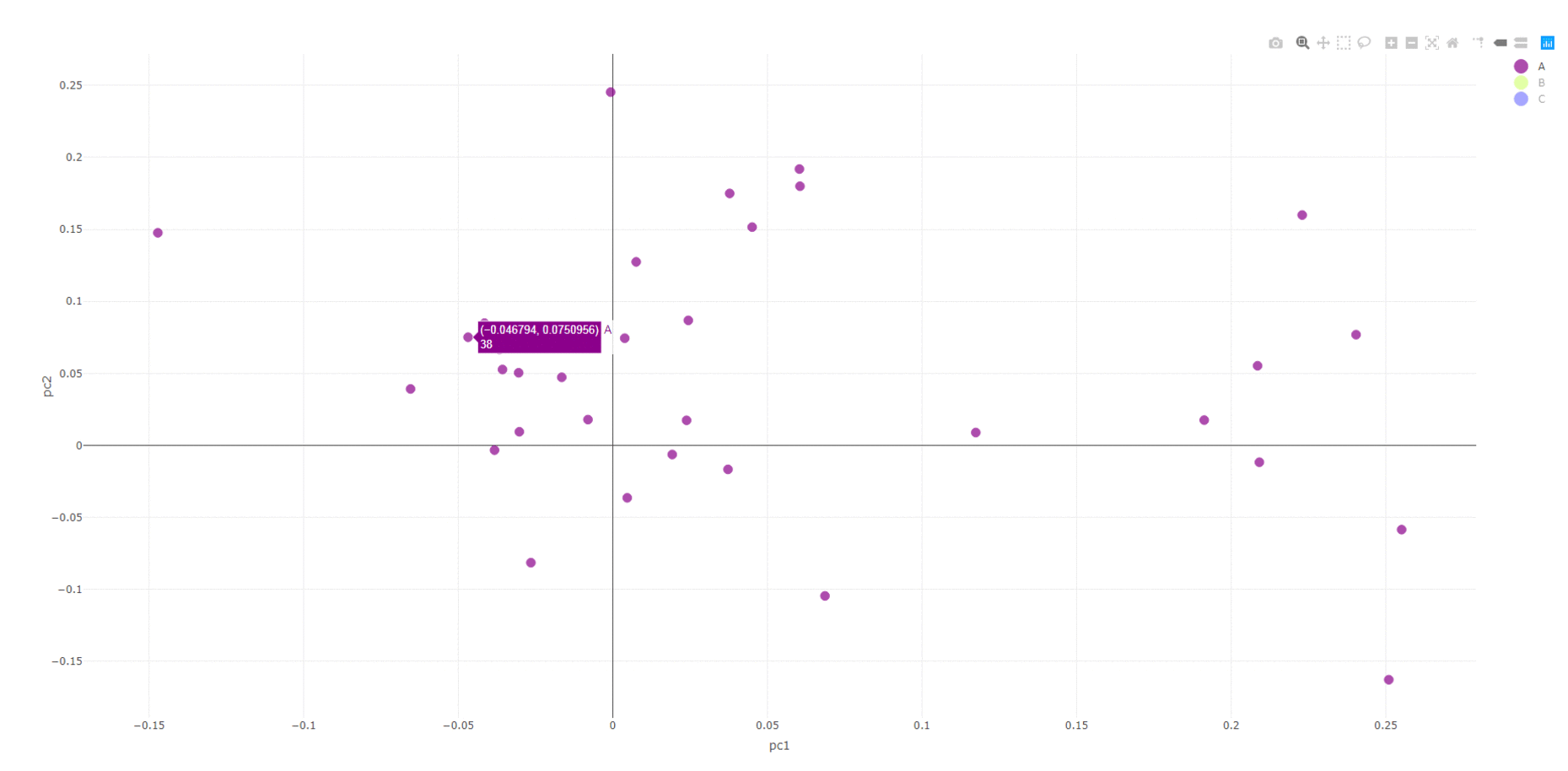
网页打开output1.html文件,如下图所示,可以选择性打开或关闭某些分组,,鼠标移动可查看样品名称。
Out[11]:
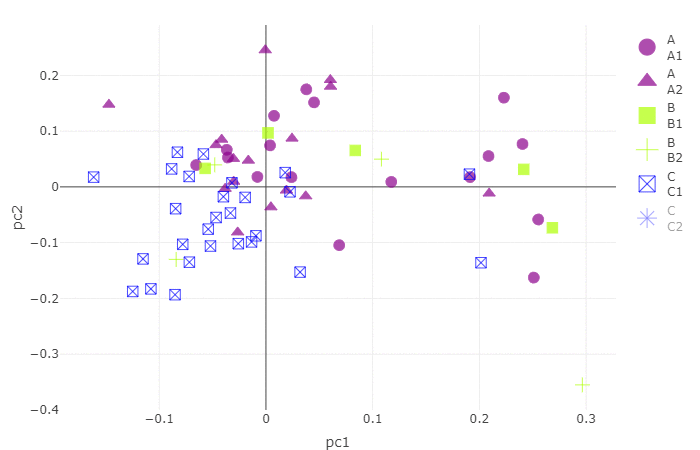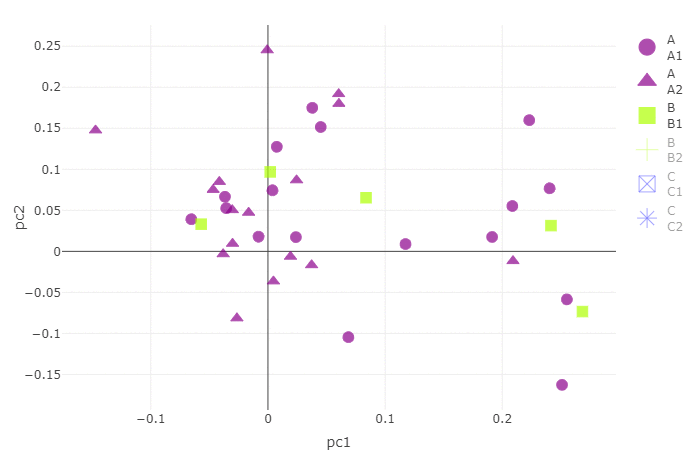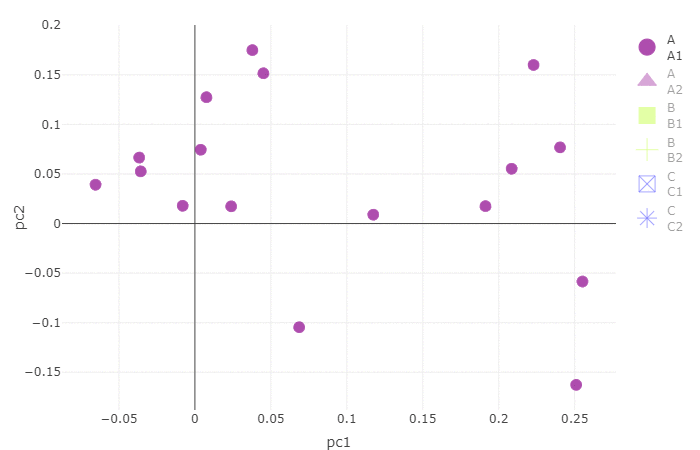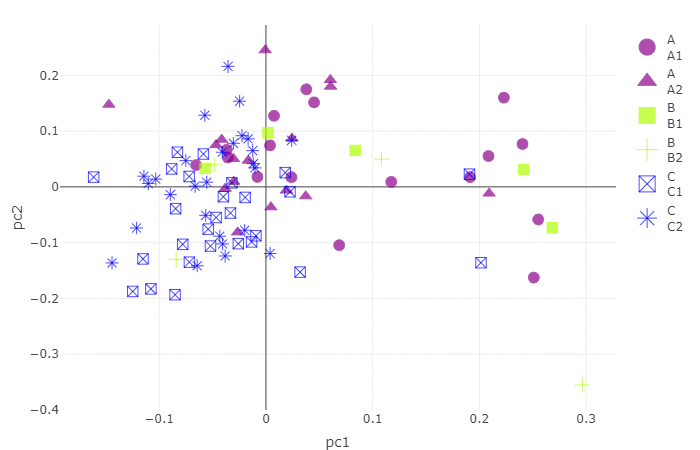
p2 = plot_ly(data_plot, type = 'scatter', x = ~pc1, y = ~pc2, color =~group,symbol =~sub_group, text =~sample, size = size, alpha = alpha, colors = color_map)
本文相关代码和文件,
可关注“上海天昊生物”公众号,
回复“PCA”自动获取!
往期相关链接:
3分钟学会CHIP-seq类实验测序数据可视化 —IGV的使用手册;
10分钟搞定多样性数据提交,最快半天内获取登录号,史上最全的多样性原始数据提交教程;
【WGS服务升级】人工智能软件SpliceAI助力解读罕见和未确诊疾病中的非编码突变;
20分钟搞定GEO上传,史上最简单、最详细的GEO数据上传攻略;
如果您对本文案介绍的方法或代码有疑问,
请扫码添加QQ群沟通
【本群将为大家提供】
分享生信分析方案
提供数据素材及分析软件支持
定期开展生信分析线上讲座

QQ号:1040471849
作者:大熊
审核:有才
来源:天昊生信团
微信扫一扫
关注该公众号
前往“发现”-“看一看”浏览“朋友在看”