
 咨询热线:400-065-6886
咨询热线:400-065-6886

 咨询热线:400-065-6886
咨询热线:400-065-6886

一
ggpubr绘制气泡图
df = read.table('phylum_taxon_abundance.xls',header = T,row.names = 1,check.names = F)head(df)1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16Proteobacteria 126573 92545 83038 75739 103697 116154 73868 76555 47648 61165 90984 86653 111958 95141 92972 102300
Actinobacteria 12759 10186 50860 32437 34212 21024 64078 52398 46109 38587 46147 46310 46780 50249 32803 29345Bacteroidetes 18428 25557 16559 22848 7046 23059 4974 8066 4984 15941 14064 12966 10931 5047 14100 27364Acidobacteria 16566 26529 6636 16079 7563 10637 8352 11404 11755 7008 8292 9372 6405 5773 10853 6738Chloroflexi 6665 11038 12520 20040 22476 4146 23314 26628 32961 30103 14238 11799 9819 15672 7421 6116Firmicutes 6033 13830 21344 3293 18031 10253 12822 5480 35773 6208 13005 20341 7953 21295 19200 12794

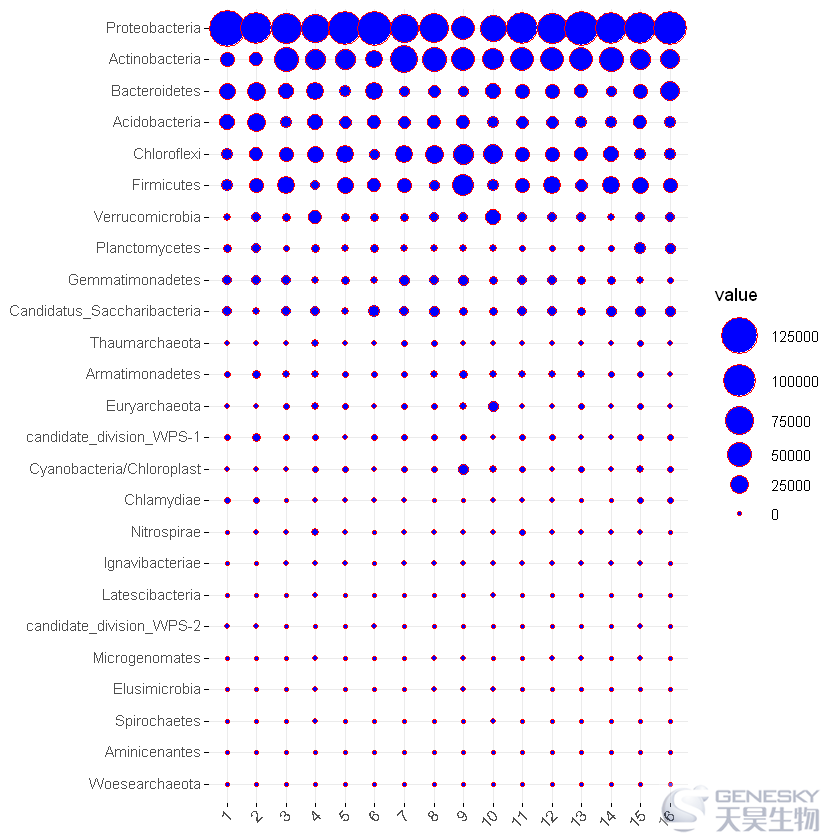
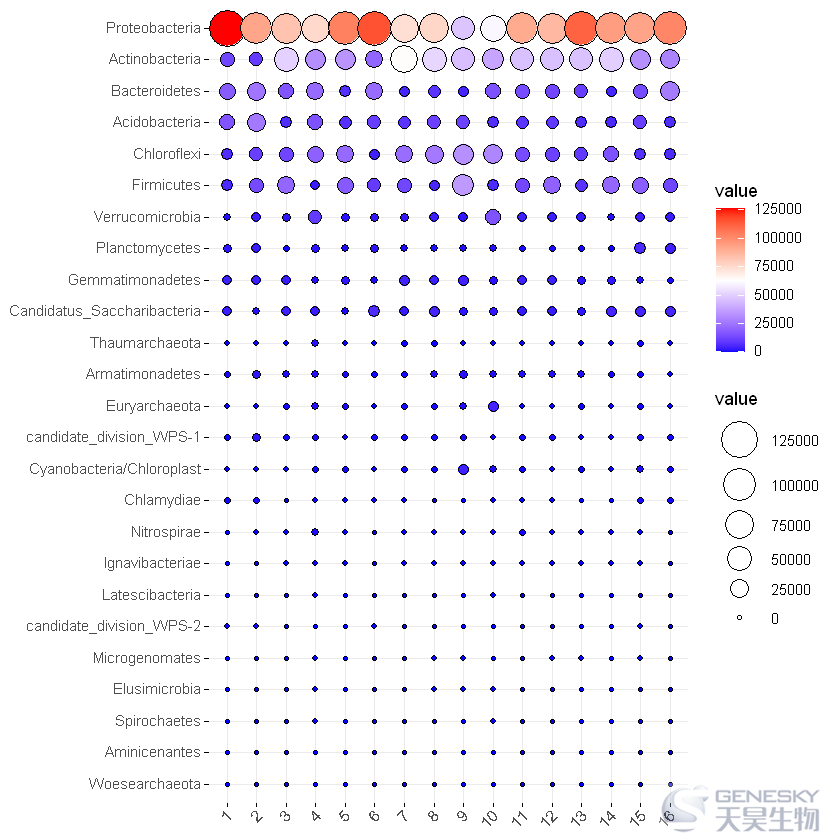
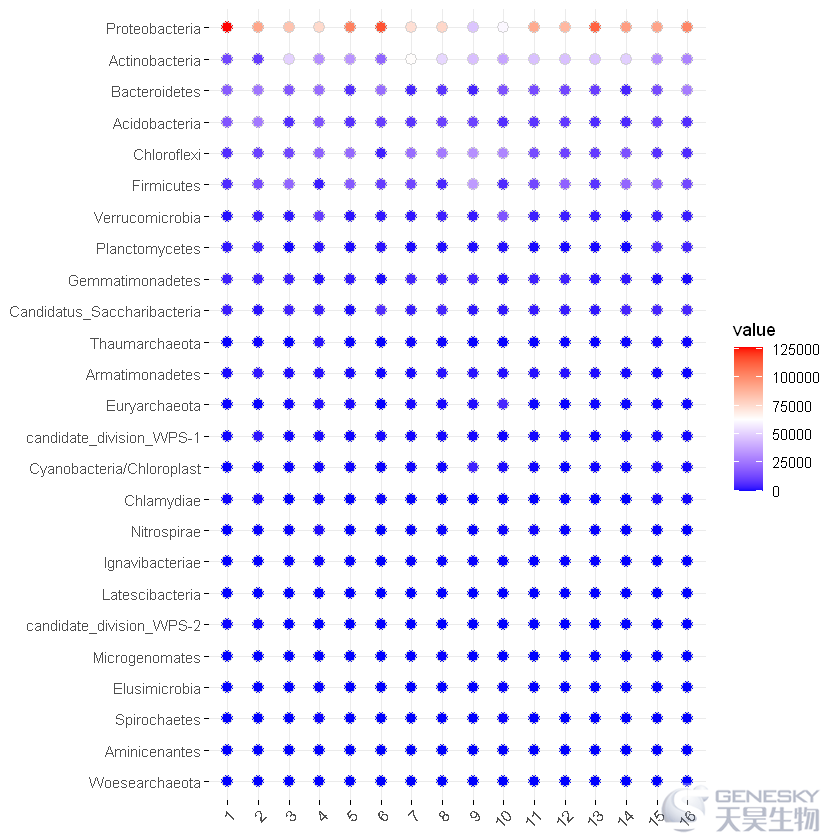
二
ggplot2绘制气泡图
data = stack(df)colnames(data) = c('Abundance','sample') head(data)Abundance sample126573 112759 118428 116566 16665 16033 1data$phylum = rep(rownames(df), times = ncol(df))head(data)Abundance sample phylum126573 1 Proteobacteria12759 1 Actinobacteria18428 1 Bacteroidetes16566 1 Acidobacteria6665 1 Chloroflexi6033 1 Firmicutesgroup = rep(c('A','B'),each = 25*16/2)length(group)Abundance sample phylum group
126573 1 Proteobacteria A12759 1 Actinobacteria A18428 1 Bacteroidetes A16566 1 Acidobacteria A6665 1 Chloroflexi A6033 1 Firmicutes Aggplot(data, aes(x = sample, y = phylum, size = Abundance, colour = group)) + geom_point() + theme_bw() +scale_size(range = c(0,6)) + labs(x = "Sample", y = "")
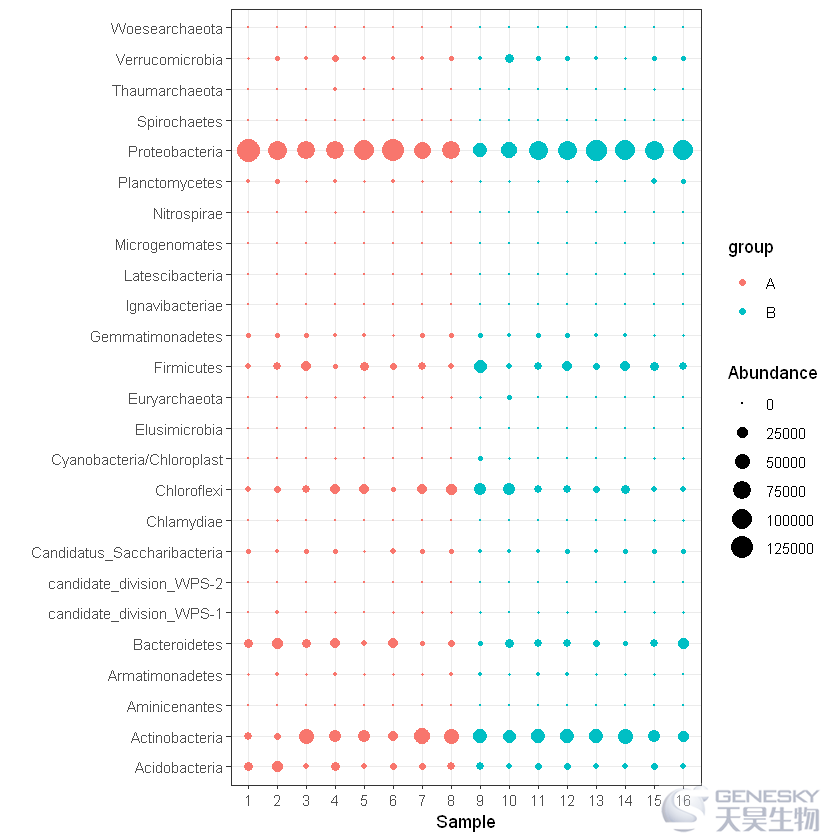
往期相关链接:
1、R基础篇
2、R进阶
【绘图进阶】之交互式可删减分组和显示样品名的PCA 图(三);
3、数据提交
3分钟学会CHIP-seq类实验测序数据可视化 —IGV的使用手册;
10分钟搞定多样性数据提交,最快半天内获取登录号,史上最全的多样性原始数据提交教程;
20分钟搞定GEO上传,史上最简单、最详细的GEO数据上传攻略;
4、表达谱分析
5、医学数据分析
【本群将为大家提供】
分享生信分析方案
提供数据素材及分析软件支持
定期开展生信分析线上讲座

QQ号:1040471849
作者:大熊
审核:有才
来源:天昊生信团
微信扫一扫
关注该公众号
前往“发现”-“看一看”浏览“朋友在看”