
 咨询热线:400-065-6886
咨询热线:400-065-6886

 咨询热线:400-065-6886
咨询热线:400-065-6886
背景
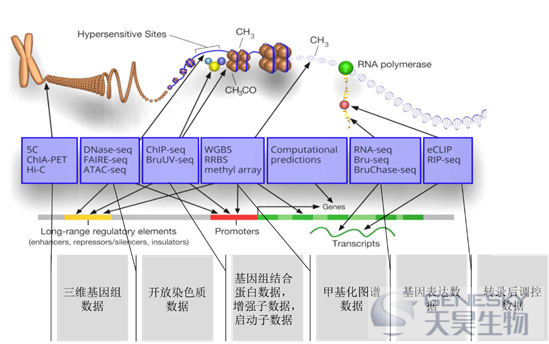
图:ENCODE计划涉及的实验内容和数据类型
HaploReg和RegulomeDB数据库,将ENCODE数据库中的调控数据与基因组中SNP数据结合起来,从而实现了对SNP调控功能的综合评价。
Haploreg数据库
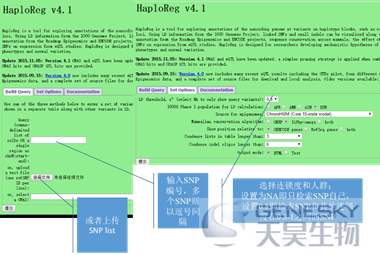
●以rs2854510为例的综合结果展示页面,每个SNP的详细注释结果需点击SNP的名字

1. SiPhy cons:序列保守性注释(SiPhy软件)。
2. Promoter histone marks,Enhancer histone markers,DNAse:来源于DNAse和ChIP-Seq实验的调控元件分类注释结果,包括是否处于组蛋白修饰位置(promoter,enhancer等);属于什么染色质状态Chromatin States(来自Roadmap项目);是否处于染色质开放区域(DHS区域)。
具体信息需要查看SNP位点详细页面的信息:第4列(完成实验的细胞系类型),第5-11列 (具体调控实验结果,黑色为数据缺失,白色为没有检测到),其它颜色代表有具体结果。

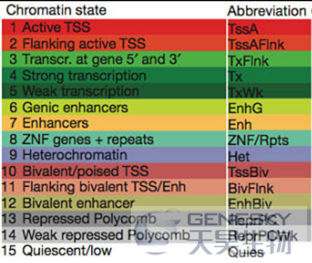
1)Chromatin states (Core 15-state model)
包括前面8个活跃状态和后面7个抑制状态,具体含义和缩写见下图。
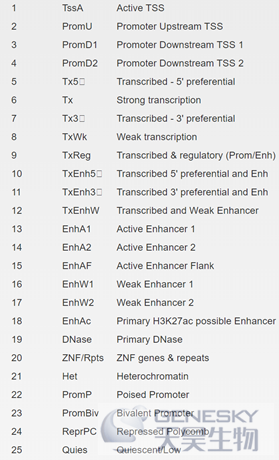
2) Chromatin states (25-state model using 12 imputed marks)
具体含义和缩写见下图。
3)H3K4me1, 与基因的转录激活有关,代表此处是转录增强子peak
4)H3K4me3, 与基因的转录激活有关,代表此处是启动子区peak
5)H3K27ac,与基因的转录激活有关,代表此处是转录增强子peak
6) H3K9ac, 与基因的转录激活有关,代表此处是启动子区peak
7) DNase,说明此处对DNaseⅠ表现出高度敏感,处于转录活跃区
3.Proteins bound :基于ChIP-Seq 实验,展示在何种细胞中SNP处于何种转录因子的结合位点.
具体信息需要查看SNP位点详细页面的信息:

4.Motifs changed:基于ENCODE 的转录因子CHIP-SEQ数据,整理了这些转录因子的基因组结合motif。这里会展示SNP的两个碱基对motif的影响。
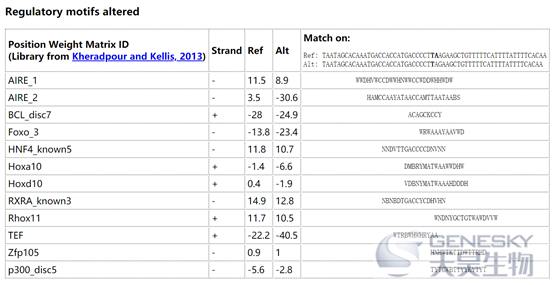
5.NHGRI/EBI GWAS hits:基于NHGRI catalog,提示该SNP是GWAS研究的阳性位点

6.GRASP QTL hits:基于GRASP数据库,提示该SNP是某个性状的阳性位点
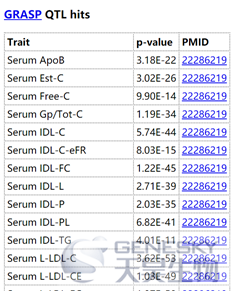
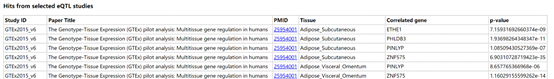
7.Selected eQTL:基于GTEx analysis V6, the GEUVADIS analysis, 和其它10篇文献,提示该SNP在XXX组织中调控XXX基因表达
RegulomeDB数据库
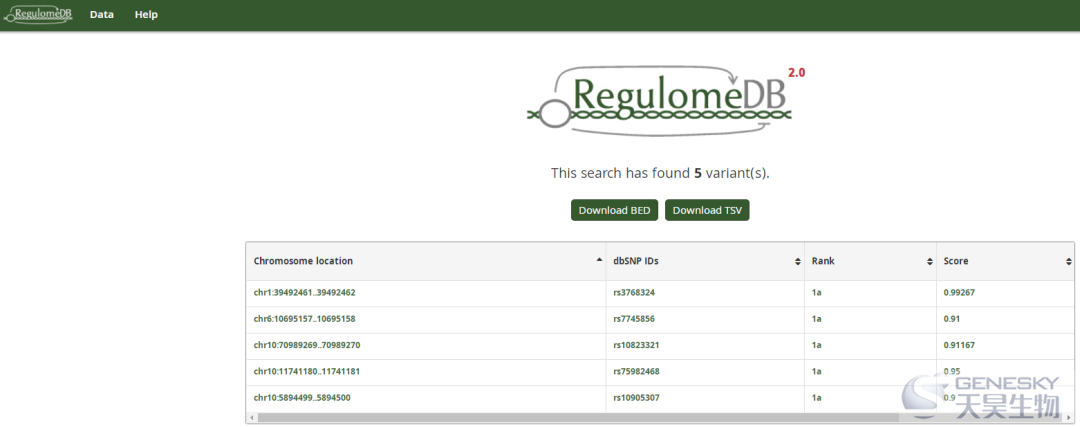

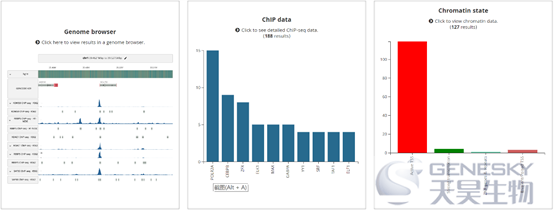
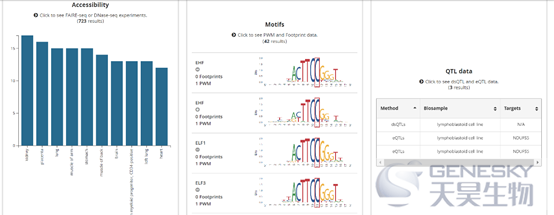
细节数据基本是和Haploreg重叠的,展示的细节略有区别,建议老师们两个数据库都进行查询和参考。
扩展阅读:
基于HaploReg和RegulomeDB数据库的生信挖掘文章:Liao, X., Lan, C., Liao, D. et al. Exploration and detection of potential regulatory variants in refractive error GWAS. Sci Rep 6, 33090 (2016). https://doi.org/10.1038/srep33090

创新基因科技,成就科学梦想
微信扫一扫
关注该公众号
前往“发现”-“看一看”浏览“朋友在看”