用于分析丛枝菌根真菌(AMF)群落组成的6对常用引物的效果比较
英文题目:Evaluation of six primer pairs targeting the nuclear rRNA operon for characterization of arbuscular mycorrhizal fungal (AMF) communities using 454 pyrosequencing
期刊名:Journal of Microbiological Methods
发表时间:2014
技术: 扩增子测序
测序平台:Roche 454 GS-FLX Titanium测序平台
材料:
选择比利时的6个苹果果园(5个商业苹果果园和1个野生苹果果园),每个果园从中随机选择三个苹果树,将根挖出来,收集已知含有丛枝菌根真菌AMF的细根。继菌根真菌的微观验证后,将根切成1–2厘米小块,用无菌蒸馏水冲洗两次,然后使用(MoBio Laboratories Inc.,Solana Beach, CA, USA) UltraClean 植物 DNA 提取试剂盒从0.25 g根部组织中提取DNA用于下一步的扩增子实验。
研究背景:
菌根是
自然界中一种普遍的植物
共生现象, 它是土壤中的
菌根真菌菌丝与高等植物营养根系形成的一种联合体。丛枝菌根真菌(AMF)(球囊菌门)与80%的陆生植物物种的根形成共生体系,丛枝菌根真菌从植物体内获取必要的碳水化合物及其他营养物质, 而植物也从真菌那里得到所需的营养及水分等,因此AMF在生态系统中发挥着关键作用,因此,量化和了解它们的分布和多样性是非常重要的。所有的丛枝菌根真菌(AMF)属于球囊菌门,它分为四个目(多孢囊霉目、球囊霉目、原囊霉目、类球囊霉目)和10个科,大部分AMF属于多孢囊霉科、无梗囊霉科、巨孢囊霉科、原囊霉科等。
内转录间隔区ITS已被建议作为真菌的标准条形码,但是这个区域它并不能区分相互之间特别紧密的物种。因此Stockinger等建议一个1500 bp区段作为真菌的标准条形码,这个区段包括一段小亚基(SSU)rRNA基因,整个的ITS区段,和一部分大亚基(LSU)rRNA基因。然而目前小亚基(SSU)rRNA基因是最常用的用于分析丛枝菌根真菌(AMF)群落的区段。目前针对SSU不同区段的引物用于分析AMF群落结构,NS31–AM1引物对是第一对用于分析AMF群落结构的引物,这个引物的缺点是能检测到非AMF物种,并且不能检测到AMF家族Ambisporaceae,Archaeosporaceae 和 Paraglomeraceae的所有物种。AMV4.5NF–AMDGR引物与NS31–AM1引物对相比,能检测到绝大部分球囊菌门物种。AML1–AML2引物在使用silico测序研究球囊菌门时具有较好的特异性和覆盖度。在焦磷酸测序时,用的是NS31–AML2引物。除了SSU区域,LSU区域也被用于分析AMF群落结构,最常用的引物对是FLR3-LSUmBr和Glo454-NDL22。因此,这项研究目标是评价以前常用于AMF研究的六个不同AMF引物(表1,图1)在AMF群落结构分析的能力:(I)获得的高质量序列的数量;(ii)AMF物种的选择性;(III)重复性;(IV)准确解析AMF群落结构的能力。
表1: 用于本文研究的六个不同AMF引物
图1:示意图显示目引物的位置。虚线表示测序区域和箭头表示测序方向。
研究结果:
目标区域评价
四个具有较高核苷酸多样性的SSU区域被发现,其中最具变化的是引物对AMV4.5NF–AMDGR扩增出的区段(图2A)。而AML1–AML2,NS31–AML2,NS31–AM1扩增出的区段包含不太可变的下游区段。在LSU区域,两个引物对(FLR3–LSUmBr和Glo454–NDL22)同时扩增出具有较高核苷酸多样性水平的片段(图2B)。
图2:丛枝菌根真菌(AMF) rRNA基因SSU和LSU的核苷酸多样性(Pi)。
引物评价
为了评估候选引物检测丛枝菌根真菌(AMF)物种的能力,候选引物被用于从球囊菌门序列数据库中筛选与之匹配的序列(图3)。获得最佳(即最低)PrimerProspector分数的是NS31 和NDL22,它们能检测广泛的真菌。而AMDGR和 AML2则针对球囊菌门具有较好的特异性几乎能匹配上所有球囊菌门序列。对于FLR3,AM1,AMV4.5NF,score《1的部分分别占到所有球囊菌门序列的90%,80%和70%。而score在1-2之间的部分主要是由于引物3’端内部或外部5bp区域的不匹配造成的,例如FLR3就是在这个区域与无柄孢子科和球囊霉科不匹配。对于AM1,则是由于在这个区域与Claroideoglomeraceae和巨孢囊霉科不匹配。对于AMV4.5NF,则是由于引物3’末端与Claroideoglomeraceae和巨孢囊霉科不匹配,或者是引物3’端序列与Glomeraceae, Pacisporaceae, Diversisporaceae。对于AML1和Glo454,score》2的部分分别占到所有球囊菌门序列的10% and 25%,这是由于引物3’末端与物种序列不匹配造成的(图3)。LSUmBr1-5引物的得分均很差。
图3:新鲜叶片(红色),生茶(蓝色)和熟茶(橙色)的普洱茶 Venn图分析
测序得到的序列数和针对AMF物种的选择性
针对样品Ha1, Ha2和Hu3,使用6对引物得到平均序列数是154,665(表3)。当序列被截断至200-250 bp时,总序列数的36.1%-92.3%能通过质量控制。当序列被截断至400 bp时,只有一小部分的序列能通过质量控制(表3)。

表3:序列截断之前和之后得到的序列数目。
通过检测高质量序列在五种主要真菌门(Ascomycota, Basidiomycota,
Chytridiomycota, Glomeromycota, Zygomycota)的分布来评估不同引物对球囊菌门(Glomeromycota)的选择性(表4)。总体来说AMV4.5NF–AMDGR、AML1–AML2、 NS31–AML2分别产生69,663、73,369、47,992条200–220bp高质量的球囊菌门序列。 使用引物AML1–AML2得到都是球囊菌门的代表序列,表明此引物具有AMF高特异性。使用引物AMV4.5NF–AMDGR 和NS31–AML2得到了球囊菌门四分之三的的代表序列。AMV4.5NF–AMDGR得到的非特异序列大部分属于擔子菌門 (11.1%)和壶菌门 (4.3%)。NS31–AML2得到的非特异序列大部分属于其它真核生物,如线虫类(表4)。引物NS31–AM1、FLR3–LSUmBr、Glo454–NDL22只得到特别少部分的球囊菌门序列,分别占总序列数的(少于1%)、(3.4%) 、(0.3%)。NS31–AM1得到大部分是子囊菌门,而FLR3–LSUmBr、Glo454–NDL22得到大部分是擔子菌門(分别占86.3%和92.0%)。无论序列是截断至200-250 bp或400bp,得到的高质量序列在五种主要真菌门的分布没有明显差异。

表4:检测6对引物得到的高质量序列在五种主要真菌门的分布
AMF群落组成、重现性和多样性
因为引物AMV4.5NF–AMDGR、AML1–AML2、 NS31–AML2能得到球囊菌门大部分的代表序列,所以进一步评价了这3对引物在分析AMF群落组成方面的差异,结果发现这三对引物得到的AMF群落组成没有显著差异(图S2)。并且使用同一对引物得到的生物学重复样本之间的AMF群落组成也没有显著性差异,表明这3对引物在分析AMF群落组成方面具有优异的重现性。
使用引物AMV4.5NF–AMDGR、AML1–AML2、 NS31–AML2分别得到AMF 33个,26个,28 个OTUs,这三对引物同时检测到的OTUs有20个(图4)。这三对引物所得到的OTUs数目相互之间没有明显的差异(图5),然而当把引物AMV4.5NF–AMDGR +AML1–AML2或AMV4.5NF–AMDGR +NS31–AML2所得到的OUT叠加时,每个样本得到的OTUs数目就会有显著增加,而AML1–AML2 +NS31–AML2所得到的OUT叠加时,所得到的OTUs数目则没有显著增加。而三对引物叠加时AMV4.5NF–AMDGR+AML1–AML2+NS31–AML2则相对于AMV4.5NF–AMDGR +AML1–AML2或AMV4.5NF–AMDGR +NS31–AML2所得到的OUT则没有显著增加。

图4:韦恩图显示引物AMV4.5NF–AMDGR、AML1–AML2、NS31–AML2共享或特异的OTUs
使用AMV4.5NF–AMDGR得到的AMF群落均匀度相对于其它单独引物对或者组合引物对显著降低(图5),表明该引物针对某些AMF物种具有扩增偏好性。
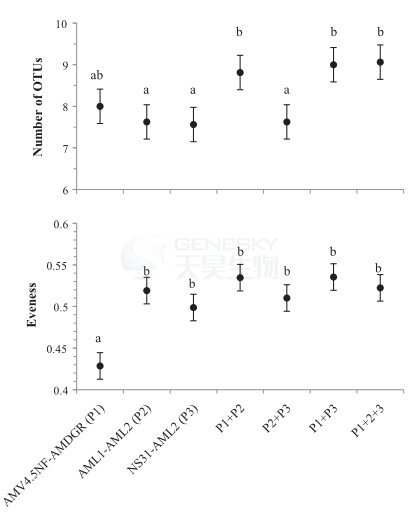
图5:使用引物AMV4.5NF–AMDGR、AML1–AML2、NS31–AML2或组合引物得到的AMF OTUs丰富度和均匀度
为了评价不同引物针对不同AMF科水平的特异性,进一步评价了使用不同引物得到的不同AMF科水平的相对丰度(表5)。AMF序列大多数属于Claroideoglomeraceae(39.93–44.04%),Gigasporaceae(12.34–12.43%)和Glomeraceae(34.95–47.39%),而ambisporaceae,Diversisporaceae和Paraglomeraceae合计占比不足11%。AMV4.5NF–AMDGR、AMV4.5NF–AMDGR + AML1–AML2、 AMV4.5NF–AMDGR + NS31–AML2能得到更多Glomeraceae序列。使用AMV4.5NF–AMDGR得到较少数目的Ambisporaceae、Diversisporaceae、Paraglomeraceae序列,尤其在鉴别Paraglomeraceae序列丰度方面,这对引物(0.11%)相对其它引物(8.84–10.07%)有明显差距。AML1–AML2 和 NS31–AML2在AMF科水平鉴定方面只有细微差别。AMV4.5NF–AMDGR引物相对于它和其它引物组合鉴定了相对较少数目的Paraglomeraceae序列和较多数目的Glomeraceae序列。

表5:使用引物AMV4.5NF–AMDGR、AML1–AML2、NS31–AML2或组合引物得到的AMF不同科水平的比例
结果总结:
1.这项研究从以下几方面评价了以前常用于AMF研究的六个不同AMF引物在AMF群落结构分析的能力:(I)球囊菌门的覆盖度;(ii)高质量的序列数;(III)AMF物种的选择性;(IV)重复性;(v)准确解析AMF群落结构的能力;
2.引物对AMV4.5NF–AMDGR、 AML1–AML2、NS31–AML2在检测高质量的球囊菌门序列数目方面(AMF的特异性和覆盖率)是优于其它3对引物的。这些引物与AMF序列只有很少的不匹配,能够始终如一地分析AMF群落结构。
3.尽管AMV4.5NF–AMDGR能够检测到较多球囊霉科(Glomeraceae)序列,但是同时得到较少数目的两性囊霉科(Ambisporaceae)、多孢囊霉科(Diversisporaceae)、类球囊霉科(Paraglomeraceae)序列。
4.引物组合AMV4.5NF–AMDGR +AML1–AML2(P1+P2),AMV4.5NF–AMDGR +NS31–AML2(P1+P2)能够检测到AMF绝大部分物种。

 咨询热线:400-065-6886
咨询热线:400-065-6886

 咨询热线:400-065-6886
咨询热线:400-065-6886









