原创 生信部 上海天昊生物
![]()
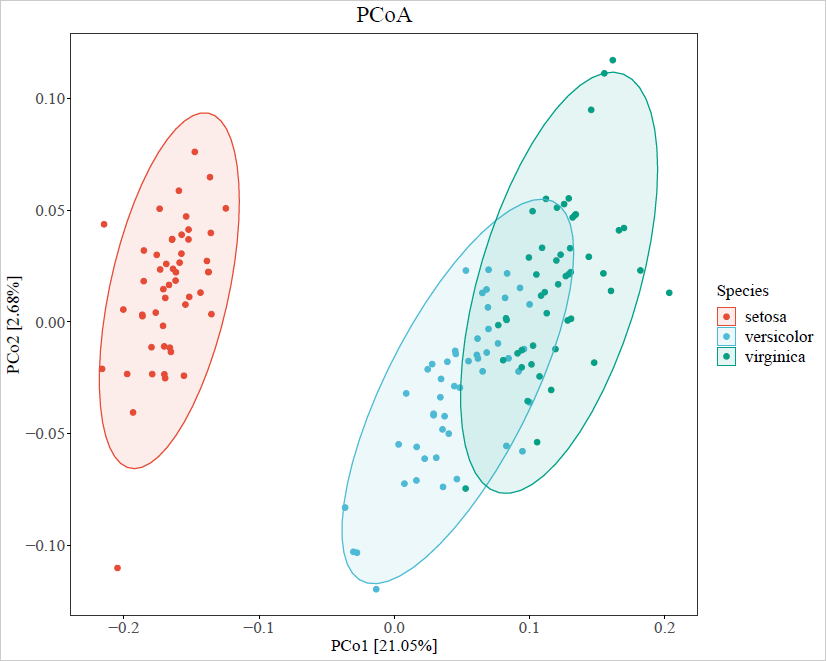
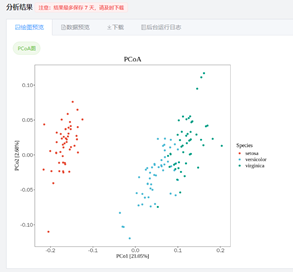
示例图
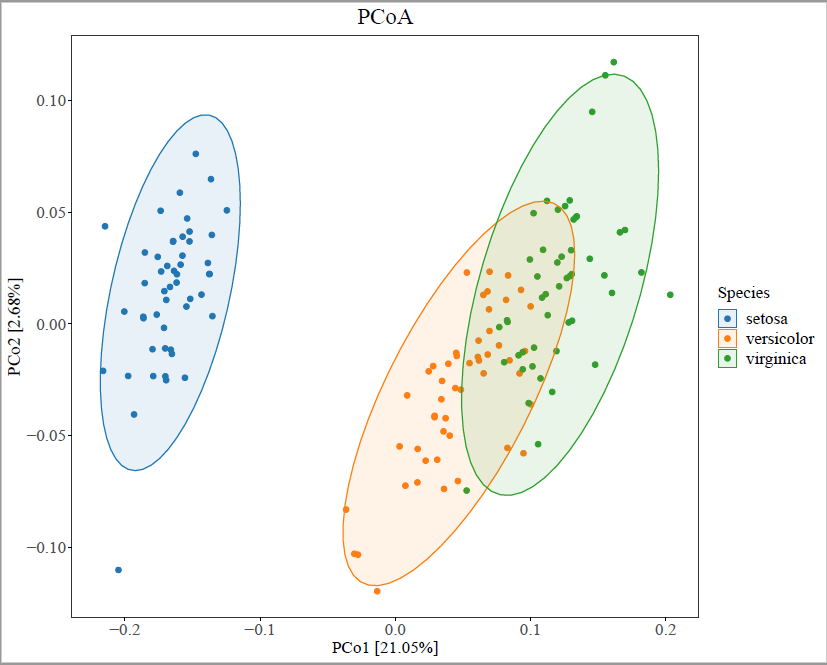
PCoA分析(Principal Coordinates Analysis)是一种非约束性的降维数据处理方法。从生物学的应用上来说,其主要目的是判断组内样本的重复性是否足够好(图上距离较近),组间样本的差异是否足够大(图上距离较远)。PCoA分析基于不同的距离算法,对一系列的特征值和特征向量进行排序,然后选择排在前几位的最主要特征值,并将其表现在坐标系里。若PCoA分析结果不好,则后续差异分析结果不可靠;若存在离群样本,可剔除该样本再进行后续分析,以确保后续结果有意义。
![]()
可以通过点击下图的“示例”按钮进行示例文件的下载,查看输入数据格式:
![]()
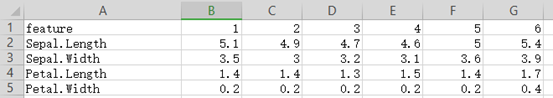
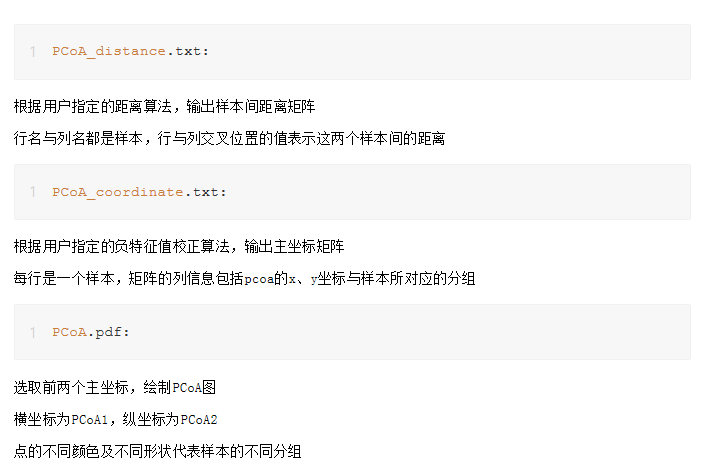
数据矩阵:文件必须是.txt后缀的UTF-8纯文本。每一行是一个特征,每一列是一个样本,列之间用制表符(Tab键 )分隔。其中,第一列是特征名称,第一行是样本名称,必须包含分组文件中所有样本。
![]()
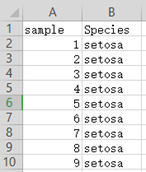
样本分组:文件必须是.txt后缀的UTF-8纯文本。第一列是样本名,用于指定需要分析的样本,第二列用于指定样本分组,第一行为表头。分组信息可用于给样本设置不同的颜色,便于区分。可以附加第三列用于指定样本在绘图中的形状。![]()
上传完成后,自动开始分析,等待十几秒即可完成。分析完成后即可预览、下载分析结果高清图。
![]()
运行完成示意图

![]()
Bray-Curtis Distance : Bray-Curtis(布雷柯蒂斯)距离取值在[0,1]之间,用来计算样本之间的差异。n维空间中的距离公式为
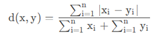
![]()
Jaccard Distance : Jaccard(杰卡德)距离用两个集合中不同元素占所有元素的比例来衡量两个集合的区分度。其距离公式为
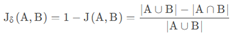
![]()
Manhattan Distance : Manhattan(曼哈顿)距离表示两个点在标准坐标系上的绝对轴距总和。其在二维数据中两点的距离为纵轴上的距离加上在横轴上的距离,即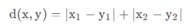
![]()
Euclidean Distance : Euclidean(欧几里得)距离表示空间中两点间的直线距离。n维空间中的欧几里得距离公式为
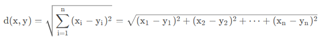
![]()
客户可自行选择是否绘制椭圆。若选择绘制,后台则会根据颜色分组,用该组颜色绘制椭圆并进行区域的划分,更加直观展现组内样本的分布情况以及不同分组件间的差异情况。客户可自行选择所需要的颜色,可以通过两种方式进行指定:(一)从所提供的不同调色板中选取,绘图颜色将会自动从该调色板中随机取出。
![]()
(二)自行指定颜色,需要使用6或8位的十六进制颜色代码,前六位表示颜色,后两位表示透明度(透明度范围从00全透明—FF不透明),6位则默认不透明。颜色个数不能少于颜色分组的个数,当指定颜色个数多于分组数时,就前读取,多种颜
![]()
![]()
输入文件必须是.txt后缀的UTF-8纯文本,列之间用制表符(Tab键 )分隔。
[1] Oksanen J, Simpson G, Blanchet F, et al. (2022) vegan: Community Ecology Package https://CRAN.R-project.org/package=vegan
[2] Paradis E, Schliep K (2019). ape 5.0: an environment for modern phylogenetics and evolutionary analyses in R, *35*, 526-528. doi:10.1093/bioinformatics/bty633
咨询沟通请联系

18964693703(微信同号)
![]()
官方网址:http://www.geneskybiotech.com

 咨询热线:400-065-6886
咨询热线:400-065-6886

 咨询热线:400-065-6886
咨询热线:400-065-6886









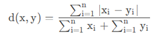
![]()
![]()
![]()

![]()

