
 咨询热线:400-065-6886
咨询热线:400-065-6886

 咨询热线:400-065-6886
咨询热线:400-065-6886
中文题目:早产儿微生物群组装的多界生态驱动因素

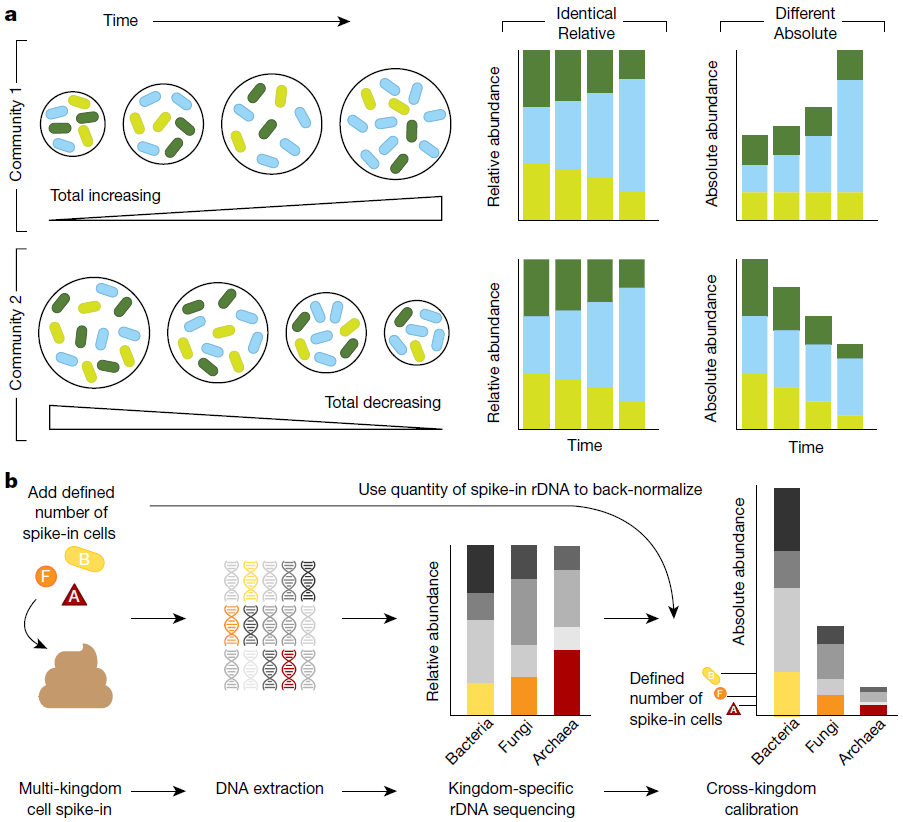
图1、MK-SpikeSeq能够对微生物绝对丰度进行有效的定量。a,示意图展示了相对丰度数据如何掩盖潜在的群落动态情况,使得区分不同的生态情景变得具有挑战性。b,MK-SpikeSeq流程概述。在DNA提取之前,将一定量的Spike-in内标菌(细菌(B)、真菌(F)和古菌(A))添加到每个样品中。然后使用标准的界水平特异性rDNA扩增子测序来进行绝对丰度的定量。由于每个Spike-in内标菌的rDNA绝对丰度是已知的,因此可当作反向归一化因子,来计算每个样品中存在的所有其他微生物的绝对丰度。添加内标菌还用作整个样品处理过程的内部对照,使绝对定量对诸如样品间DNA提取效率差异等因素具有鲁棒性。
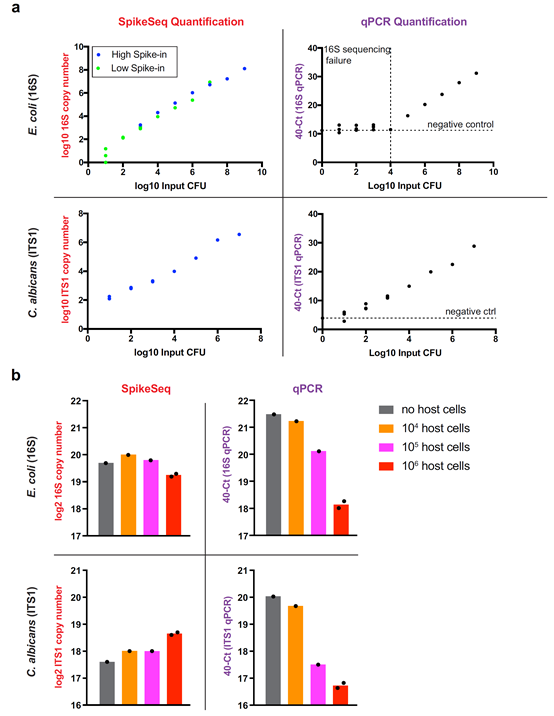
图2、MK-SpikeSeq在检测灵敏度和对样品背景的鲁棒性方面优于qPCR。a,使用Escherichia coli和C. albicans的10倍梯度稀释,比较MK-SpikeSeq和qPCR之间的灵敏度。MK-SpikeSeq结果显示,在低细菌丰度样品中,相对于qPCR的灵敏度提高了约100-1000倍(仅检测10个细菌细胞)。对于E. coli样品的MK-SpikeSeq,使用两个梯度的Spike-in内标来覆盖每个样品约10000-100000个读序测序深度的整个检测范围。对于qPCR,水平虚线表示阴性对照(水DNA),垂直虚线表示下限阈值。低于该阈值时,16S测序产生少于100个读序(测序失败,可能是由于信号太低)。b,使用含有一定量E. coli和C. albicans以及可变数量 Caco-2 结肠细胞的测试样品,比较 MK-SpikeSeq 和 qPCR 对宿主细胞背景的稳健性。MK-SpikeSeq在具有高宿主细胞背景干扰的样品中检测到一致的(<2倍变化)微生物丰度,而qPCR方法在不同宿主细胞背景干扰的微生物丰度检测结果差别高达10倍以上(ΔCt> 3.3)。

1
天昊生物Accu16S®细菌和AccuITSTM真菌绝对定量测序专利技术简介:
天昊生物开发的微生物Accu16S®细菌与AccuITSTM真菌绝对定量测序技术,就是一种基于添加内标序列的微生物绝对定量检测方法。本技术通过向样品DNA中添加天昊专利的人工合成已知拷贝数内标序列,然后进行16S扩增子文库构建、测序,再根据内标序列reads数及其绝对拷贝数绘制出标准曲线,最后可获得样品中OTU代表序列对应的细菌物种绝对拷贝数。利用该方法,我们可以同时获得绝对定量分析结果、常规扩增子相对定量结果,以及相对定量与绝对定量的比较分析结果,可谓“一次检测,三套结果”,数据更加全面准确。
2
天昊扩增子绝对定量测序技术优势:
一次检测,三套数据,结果更丰富;
对样本的需求量要求低,避免因样本无备份等造成的无法检测情况;
检测通量高,检测合格范围内各类菌种均可绝对定量;
相对定量和绝对定量数据相互印证,减少传统相对定量假阳性问题;避免跨平台qPCR定量的系统误差;
内标法比qPCR在定量上具有更高的特异性、灵敏度和更好的一致性;
避免因样本DNA抽提等原因造成的PCR抑制剂残留对结果准确性的影响;
避免qPCR等定量实验碰到的引物设计和优化难题。
截至目前,天昊客户利用微生物绝对定量测序共发表SCI文章42篇,其中JCR分区在Q1区的高分文章多达25篇,研究领域涉及医学、环境、食品、农学和生态等各个领域,样本类型包括人类及动物粪便、土壤、体外发酵液、水体生物膜、酒类发酵物及口拭子DNA样本等。


往期相关链接:

官方网址:http://www.geneskybiotech.com