
 咨询热线:400-065-6886
咨询热线:400-065-6886

 咨询热线:400-065-6886
咨询热线:400-065-6886

示例图
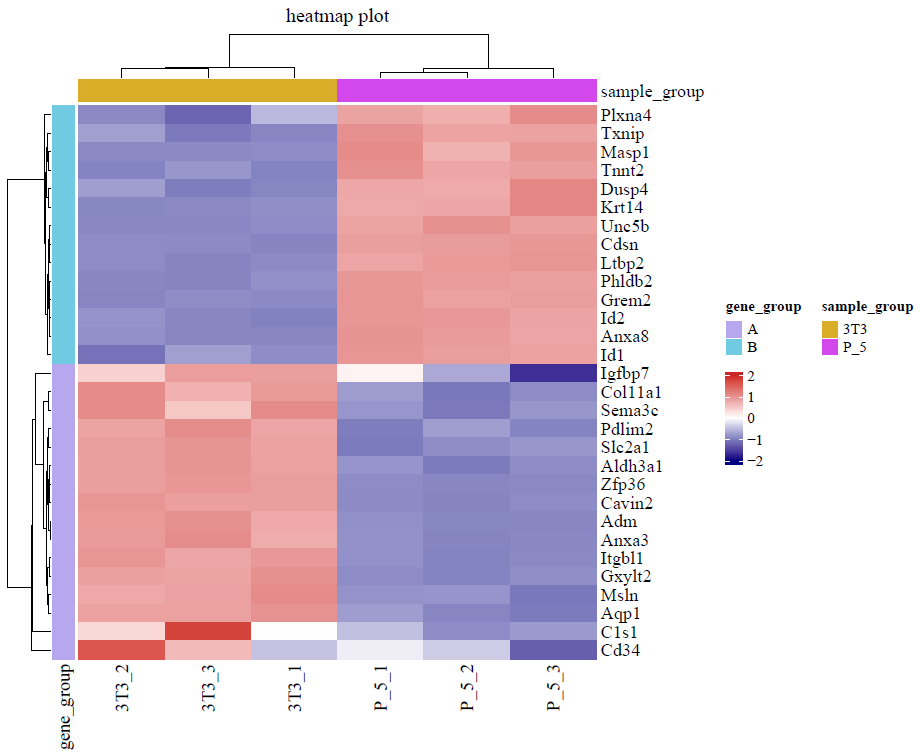
热图是由多个小色块组合而成,每一个小色块都代表一个数值,根据给定色彩变化尺给每一个数值色块分配颜色。
数值可能会有较大差异,跨越几个数量级,因此通常会给数值取对数或者Z-score,使色彩变化尺处于个位数。
聚类是利用多组值间两两的差异程度或者相似程度作为依据,对多组值进行层析聚类,以最终得到样本间聚类的远近关系。
一般,样本来自于同一个组,组内样本间的特征相似,在进行聚类的时容易被聚在一起。
基因聚类可以观察到基因之间的关系,因为基因的上下游调控会导致连锁反应,一个基因的表达增加或减少可能会带动一系列基因的表达变化。


可以通过点击下图的“示例”按钮进行示例文件的下载,查看输入数据格式:

云平台数据输入
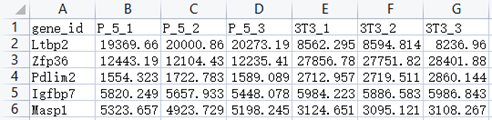
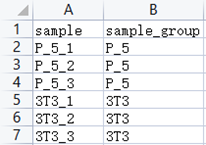
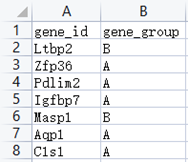

(以示例数据为例)
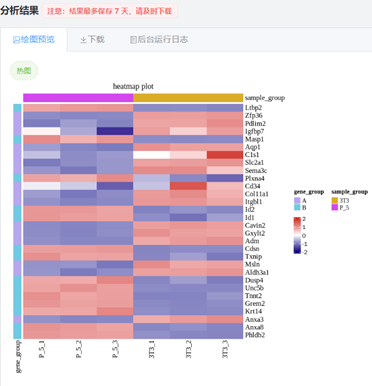
运行完成示意图

运行结果一览
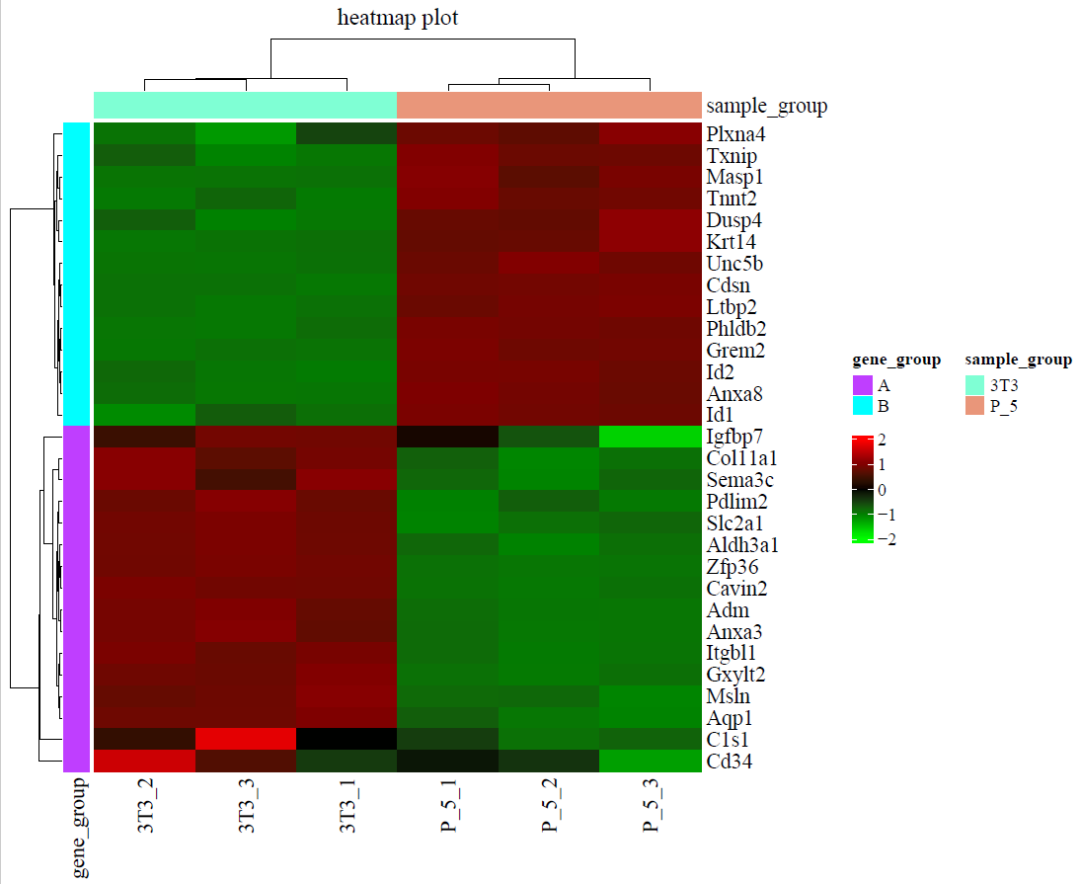
heatmap.pdf:
上方树形图为样本聚类结果,左侧树形图为特征聚类结果

正如我们所知,热图是利用颜色变化来直观地展示数值的变化。但如果绘制热图的数据差异过大,那么在绘制热图时,两个特征在不同样本中表达的微小变化很难通过颜色反映出来。所以,在绘制热图前,我们通常会对表达数据进行归一化。我们提供了三种归一化方式:
none: 不对数据进行归一化处理
row: 按行进行数据归一化
column: 按列进行数据归一化
一般来说,一个特征在不同样本间的表达分布会在热图的行方向上进行展示,所以为了展示一个特征在不同样本间的分布,我们选择按行进行归一化,即row。
5.2聚类参数
当需要观察样本/特征如何分类,相关关系如何,那么可以选择聚类。我们提供的选择有:仅在样本水平聚类,仅在特征水平聚类或两者都进行。
样本聚类,即列聚类,可以观察采集的不同组别的样本是否被分类到一起。
特征聚类,即行聚类,可以观察具有相似表达模式的基因集群。
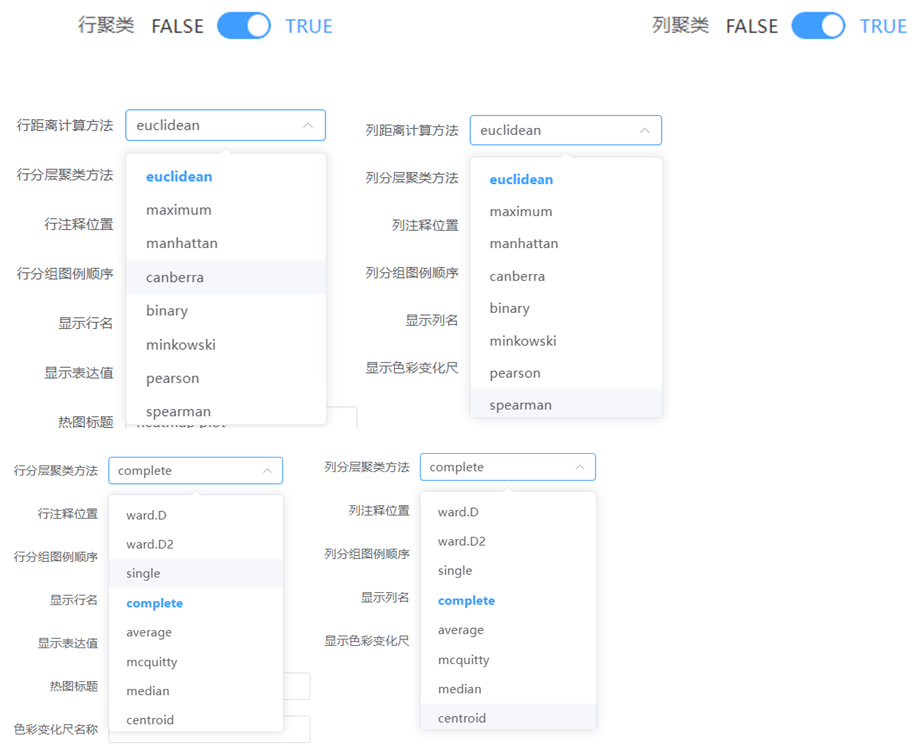
同时,如果选择聚类,我们提供了多种行/列距离算法及行/列分层聚类算法,可根据实际需求进行选择。
热图工具共提供9种距离算法:欧氏距离(euclidean), 最大距离(maximum), 曼哈顿距离(manhattan), Canberra 距离(canberra), 二进制距离(binary),明可夫斯基距离 (minkowski), 皮尔逊相关系数(pearson), Spearman 相关系数(spearman), Kendall 相关系数(kendall)。
热图工具共提供8种聚类算法:ward最小方差法(ward.D、ward.D2)、最短距离法(single)、最长距离法(complete)、类平均法(average)、相似法(mcquitty)、中间距离(median)、重心法(centroid)。
如果预先设定好的样本排序或特征排序具有生物学意义,并且想在最终的图片中呈现,通常选择不聚类。
5.3分组图例显示顺序
5.4配色的指定
![]()

输入文件必须是.txt后缀的UTF-8纯文本,列之间用制表符(Tab键 )分隔。

往期相关链接:
1、天昊云
2、R基础篇
3、R进阶
【绘图进阶】之六种带中心点的PCA 图和三维PCA图绘制(四);
【绘图进阶】之交互式可删减分组和显示样品名的PCA 图(三);
4、数据提交
3分钟学会CHIP-seq类实验测序数据可视化 —IGV的使用手册;
10分钟搞定多样性数据提交,最快半天内获取登录号,史上最全的多样性原始数据提交教程;
20分钟搞定GEO上传,史上最简单、最详细的GEO数据上传攻略;
5、表达谱分析
6、医学数据分析

官方网址:http://www.geneskybiotech.com