
 咨询热线:400-065-6886
咨询热线:400-065-6886

 咨询热线:400-065-6886
咨询热线:400-065-6886
文章导读
一
数据准备
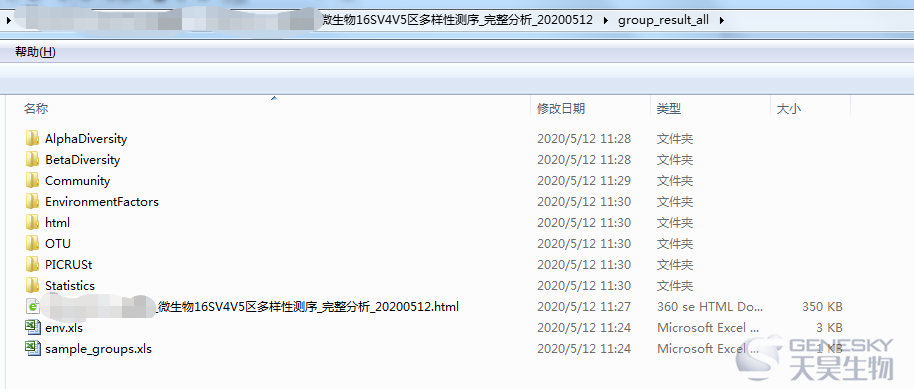
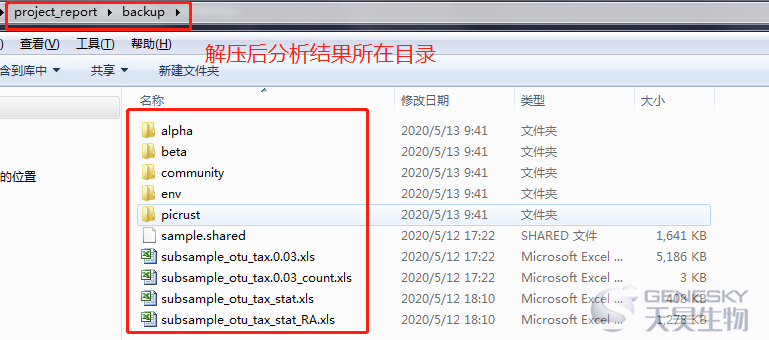
数据分析完成后,我们会将分析结果以邮件的形式发送给客户。解压后如上图所示。云平台分析所需的文件均在其中。
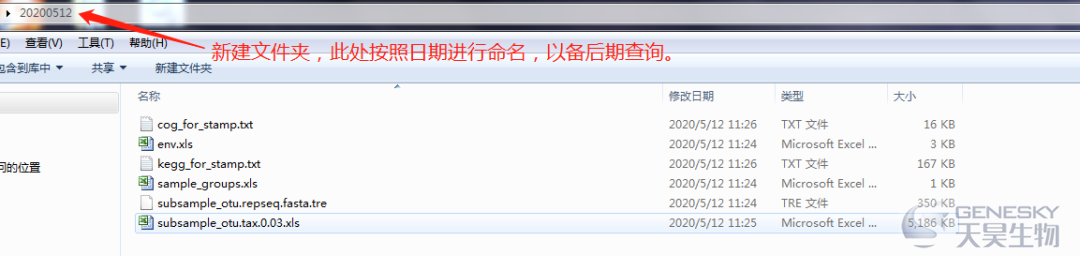
二
数据修改
1. 修改分组名(最常见)
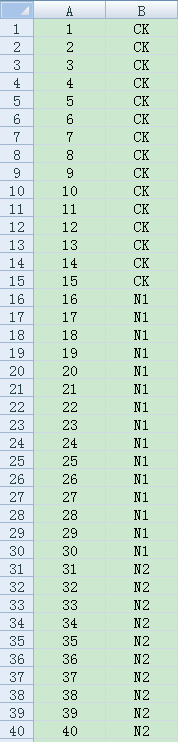
打开sample_groups.xls,A列为样品名,B列为组名。直接修改B列组名后保存并关闭,用记事本方式打开看是否乱码。或者修改后复制到新建的记事本文档中,将该新建的文档作为上传的分组文件。
2. 修改样品名(比较少见)
除subsample_otu.repseq.fasta.tre文件外,其他文件均需要修改。修改后的文件仍需要是文本文件。
三
数据提交
1. 登录
http://cloud.geneskybiotech.com/login.html ,账号密码获取,可联系qq管理员(文末扫二维码入群)。
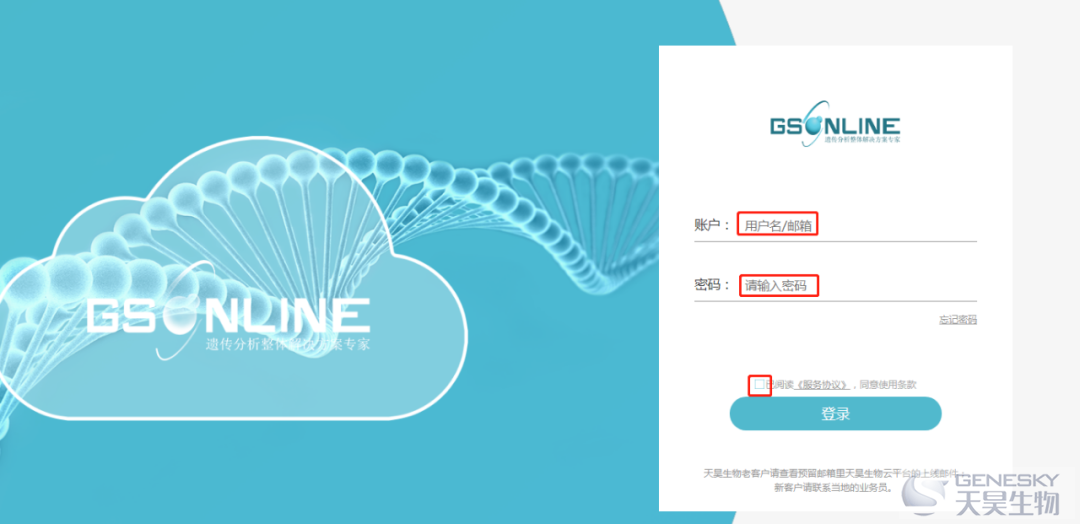
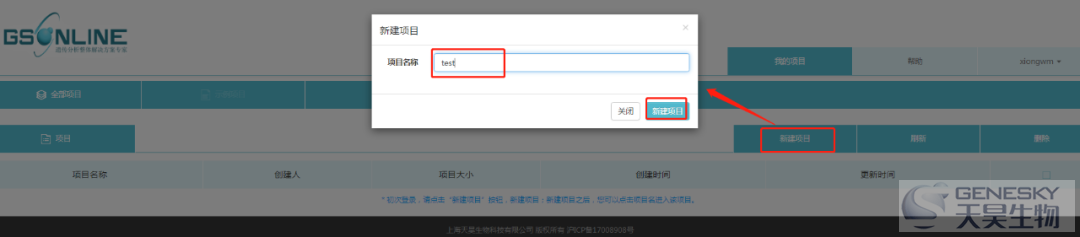
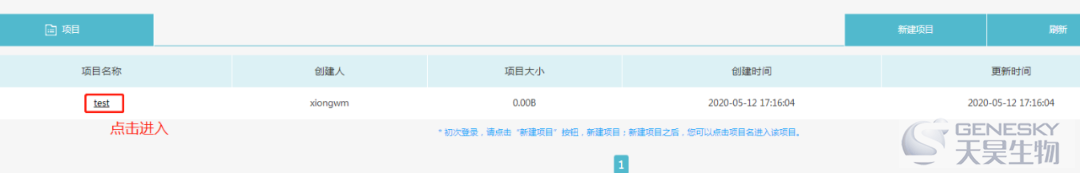
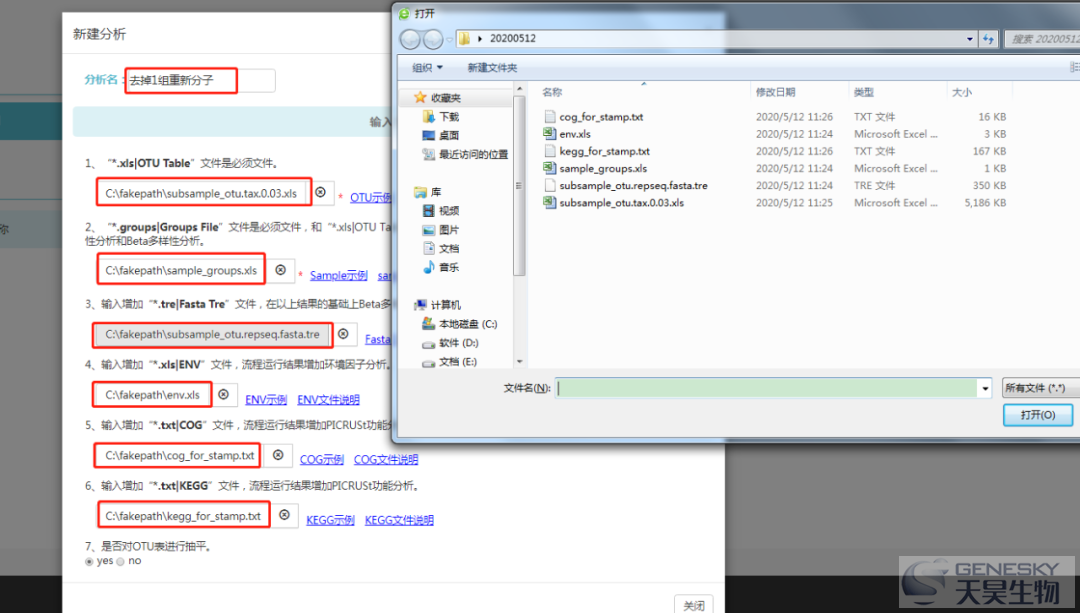
填写分析名称,从本地上传数据,点击运行。
四
查看运行与数据下载
1. 查看进展
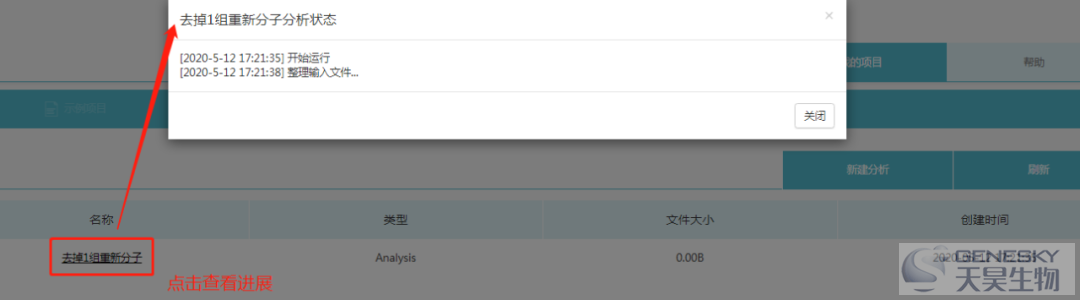
如出现其他情况,查看输入数据是否是文本格式。
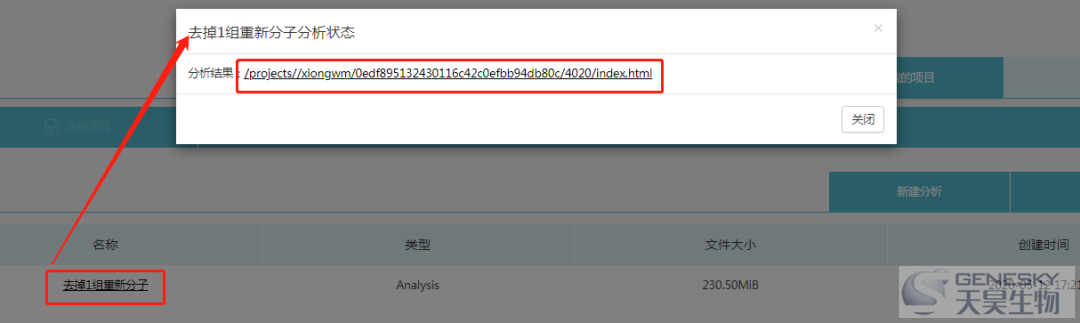

3. 结果查看
.
END
.
往期链接:
如果您对本文案介绍的方法或代码有疑问,
请扫码添加QQ群沟通
【本群将为大家提供】
分享生信分析方案
提供数据素材及分析软件支持
定期开展生信分析线上讲座

QQ号:1040471849
作者:大熊
审核:有才
来源:天昊生信团

创新基因科技,成就科学梦想
微信扫一扫
关注该公众号
前往“发现”-“看一看”浏览“朋友在看”