
 咨询热线:400-065-6886
咨询热线:400-065-6886

 咨询热线:400-065-6886
咨询热线:400-065-6886
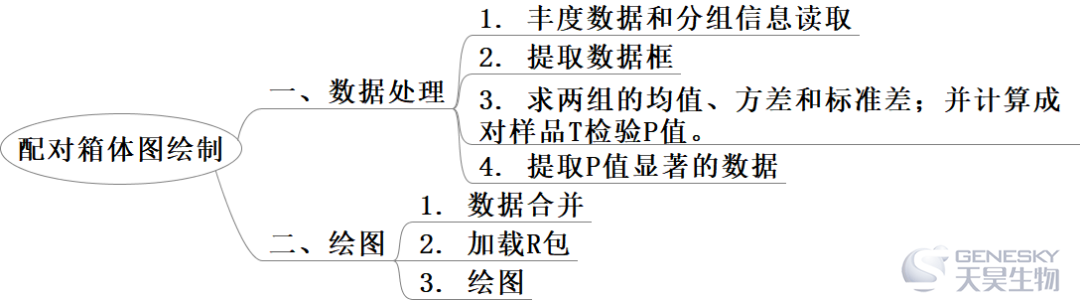

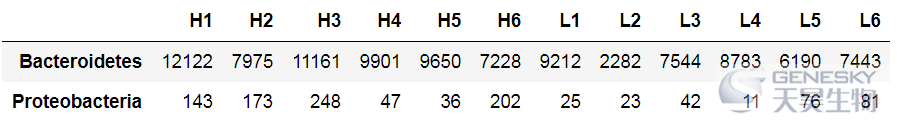
data = read.table('phylum.taxon.Abundance.xls',header = T,sep = ' ',row.names = 1)data
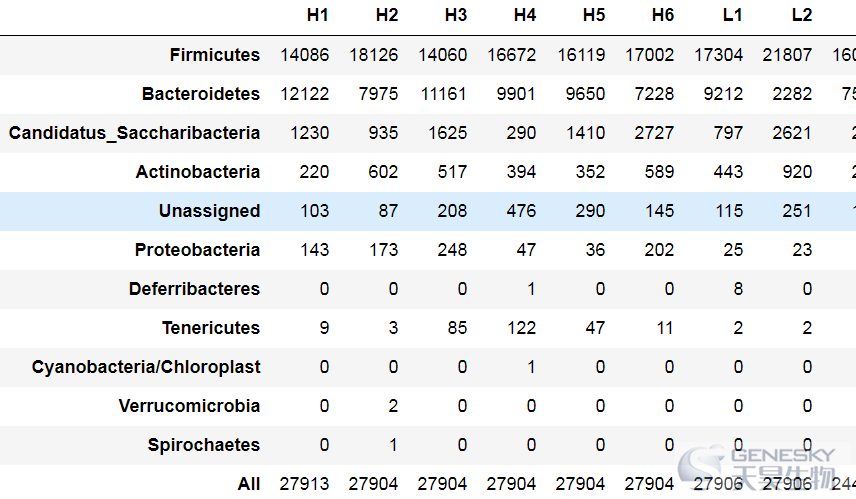
In [2]:
info = read.table('sample_groups.xls',header = F,sep = ' ',col.names = c('sample','group'))
info
Out[2]:

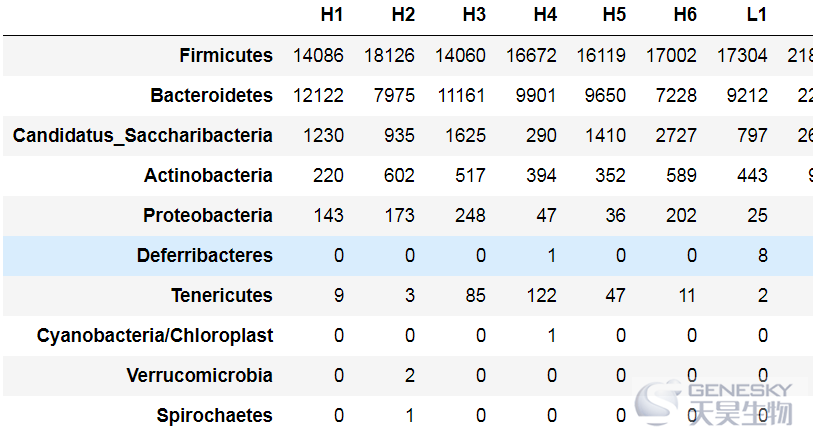
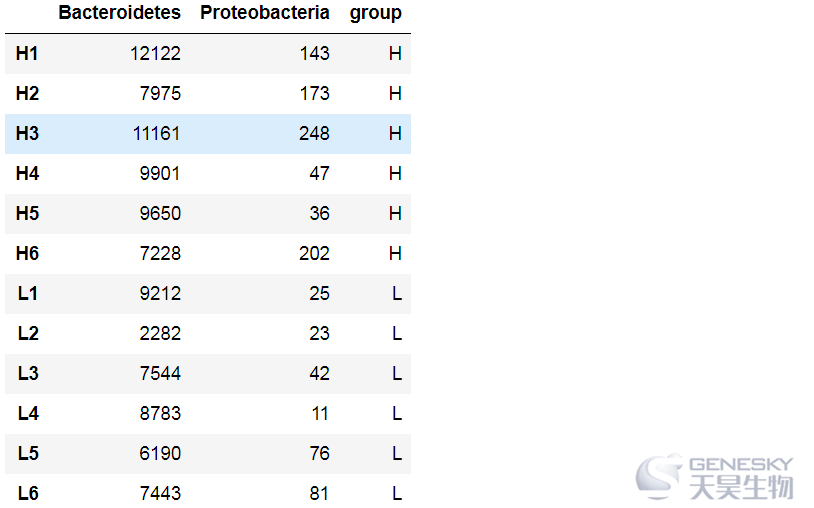
df = data[-grep("All|Unassigned", rownames(data)),info$sample]df
df['H_mean'] = apply(df[,info[info$group=='H',1]],1,mean)df['H_var'] = apply(df[,info[info$group=='H',1]],1,var)df['H_sd'] = apply(df[,info[info$group=='H',1]],1,sd)df['L_mean'] = apply(df[,info[info$group=='L',1]],1,mean)df['L_var'] = apply(df[,info[info$group=='L',1]],1,var)df['L_sd'] = apply(df[,info[info$group=='L',1]],1,sd)df['p.value'] = apply(df,1,function(x){ t.test(as.numeric(x[info[info$group=='H',1]]),as.numeric(x[info[info$group=='L',1]]),paired = T)$p.value })df
Out[5]:
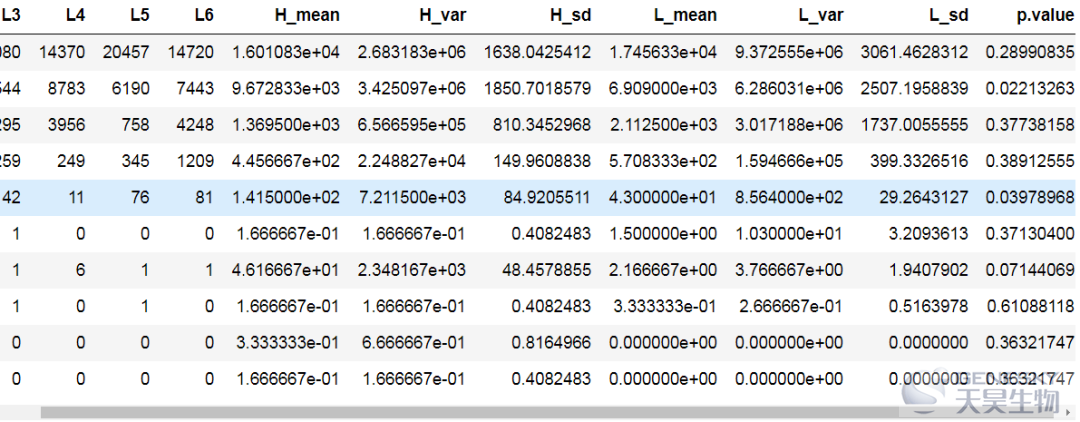
数据保存至文本文件phylum.taxon.Abundance.new.xls 中。
In [6]:
write.table(df,'phylum.taxon.Abundance.new.xls',sep = ' ',row.names = T, col.names = NA,quote = F)
4. 提取P值显著的数据
In [7]:
df_diff = df[df$p.value<0.05,info$sample]
df_diff
Out[7]:

1. 数据合并
In [8]:
df_diff = data.frame(t(df_diff),group = info$group)
df_diff
Out[8]:
2. 加载R包
In [9]:
.libPaths("C:/Program Files/R/R-3.6.1/library")
library(ggpubr)
3. 绘图
method包括:t.test、wilcox.test、anova、kruskal.test;本次分析采用t.test。
ggarrange可以将多个图绘制在一张图上,ncol=2 按照每行2张图绘制,行不限制。
In [10]:
p1= ggpaired(df_diff, x="group", y="Bacteroidetes", color = "group", line.color = "gray",xlab = 'Bacteroidetes',
line.size = 0.4, palette = "npg")+ stat_compare_means(method = "t.test",paired = TRUE)
p2= ggpaired(df_diff, x="group", y="Proteobacteria", color = "group", line.color = "gray", xlab = 'Proteobacteria', line.size = 0.4, palette = "npg")+ stat_compare_means(method = "t.test",paired = TRUE)
ggarrange(p1, p2, ncol = 2,common.legend = T)
Out[10]:

关注“上海天昊生物”公众号,
回复关键字“箱体图”,
获取文章链接,数据和代码的百度云盘链接。
往期相关链接:
1、R基础篇
2、R进阶
【绘图进阶】之六种带中心点的PCA 图和三维PCA图绘制(四);
【绘图进阶】之交互式可删减分组和显示样品名的PCA 图(三);
3、数据提交
3分钟学会CHIP-seq类实验测序数据可视化 —IGV的使用手册;
10分钟搞定多样性数据提交,最快半天内获取登录号,史上最全的多样性原始数据提交教程;
20分钟搞定GEO上传,史上最简单、最详细的GEO数据上传攻略;
4、表达谱分析
5、医学数据分析
【本群将为大家提供】
分享生信分析方案
提供数据素材及分析软件支持
定期开展生信分析线上讲座

QQ号:1040471849
作者:大熊
审核:有才
来源:天昊生信团
微信扫一扫
关注该公众号
前往“发现”-“看一看”浏览“朋友在看”