
 咨询热线:400-065-6886
咨询热线:400-065-6886

 咨询热线:400-065-6886
咨询热线:400-065-6886
10x单细胞转录组测序
全转录组测序
常规mRNA测序
常规lncRNA测序
常规circRNA测序
常规miRNA测序
微量mRNA测序
外泌体miRNA测序
RNA表达水平定量
circRNA验证
线性消化circRNA测序
全基因组甲基化测序
850K甲基化芯片检测
多重目的区域甲基化富集测序
m6A修饰整体水平定量
m6A RNA甲基化测序
m6A修饰表达水平定量
染色质开放性测序(ATAC-seq)
肿瘤全基因组测序
肿瘤全外显子组测序
肿瘤目的区域捕获测序
肿瘤泛癌种基因panel突变检测
多重目的区域甲基化富集测序
肿瘤微卫星不稳定性分析
肿瘤TCGA等数据库个性化生信挖掘
Accu16S®细菌绝对定量测序
AccuITSTM真菌绝对定量测序
常规微生物16S/18S/ITS扩增子测序
微生物功能基因扩增子测序
宏基因组测序
线粒体/叶绿体基因组测序
SSRseq®超高通量SSR分型技术
动植物目标区域测序
动植物单细胞转录组测序
动植物目的区域甲基化富集测序
Taqman SNP分型
RFLP SNP分型(mf-RFLP)
Mulitiplex Snapshot多重SNP分型
iMLDR®多重SNP分型技术
SNPscan®高通量SNP分型技术
SNPseq®超高通量SNP分型技术
SNaPshot AEI分析
GWAS芯片服务
定量PCR检测拷贝数
MLPA试剂盒检测
AccuCopy®多重DNA拷贝数检测技术
CNVplex®高通量DNA拷贝数检测技术
CNVseq®超高通量DNA拷贝数检测技术
低深度全基因组测序
人线粒体拷贝数qPCR检测
STR细胞株鉴定
候选区域排查(致病基因排查)
单基因疾病家系定位
微卫星杂合性缺失分析(LOH)
微卫星不稳定分析(MSI)
Sanger测序基因突变位点检测
Sanger测序未知序列拼接
BSP直接测序
Elisa检测
人染色体端粒长度检测
生物信息学个性化分析
非靶向代谢组
靶向代谢组
试剂与产品
原创 上海天昊生物
肿瘤微环境(TME)已被证明在各种适应症中强烈影响癌症患者的治疗结果,并影响总生存期。然而,在胃癌中形成TME的细胞还未被广泛地描述。2022年12月22日,Genome Biology杂志(IF:17.906)在线发表了BIOPIC张泽民、北京大学肿瘤医院步召德、北京大学人民医院申占龙及拜耳公司Helge G. Roider合作的论文“Parallel single‑cell and bulk transcriptome analyses reveal key features of the gastric tumor microenvironment”,利用并行的单细胞和常规转录组分析揭示了胃肿瘤微环境的关键特征,为胃癌患者分层的新潜在生物标志物的识别提供了独特的资源,有助于进一步阐明胃癌的发生机制,并为治疗提供思路。


为了表征胃癌TME,研究人员对24例未经治疗的胃癌患者肿瘤及配对的非恶性胃组织进行了bulk组织RNA测序(RNA-seq)、全外显子测序以及单细胞RNA-seq(图1A)。为了识别组成胃TME的细胞类型,样本被快速消化成单细胞,通过荧光激活细胞分选(FACS)去除EPCAM阳性的上皮细胞,富集非上皮起源的所有细胞用于单细胞分析。结果获得的96623个细胞可以分成11个主要细胞类型及81个亚型(图1B),主要细胞类型可以根据定义的标记基因进行注释(图1C)。为了在后续表征TME和正常胃基质之间的差异,作者量化了每种基质细胞类型中与恶性转化相关的细胞丰度和细胞活性的变化。
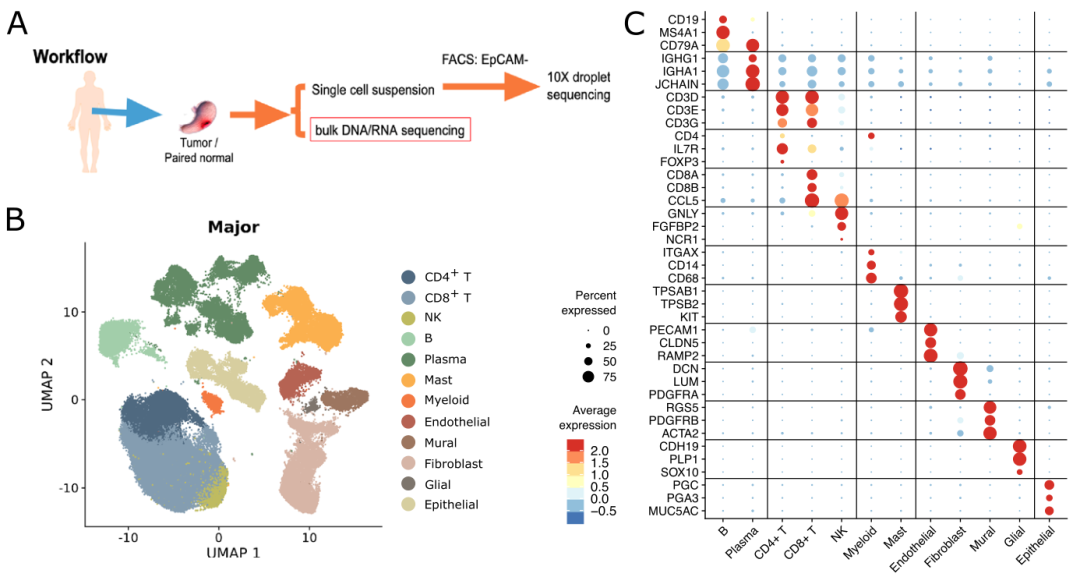
图1 良性和恶性胃样本中EPCAM阴性细胞的细胞图谱

图2 癌症相关成纤维细胞的转录组重编程
鉴于血管生成对肿瘤持续生长至关重要,作者推测这些细胞类型可能与生存率差有关,并发现在TCGA独立胃癌队列中,SERPINE1表达与生存率急剧恶化有关(图3D)。同时使用EN10-SERPINE1簇的20个最特异性基因的特征发现了生存率的可较性差异(图3E)。
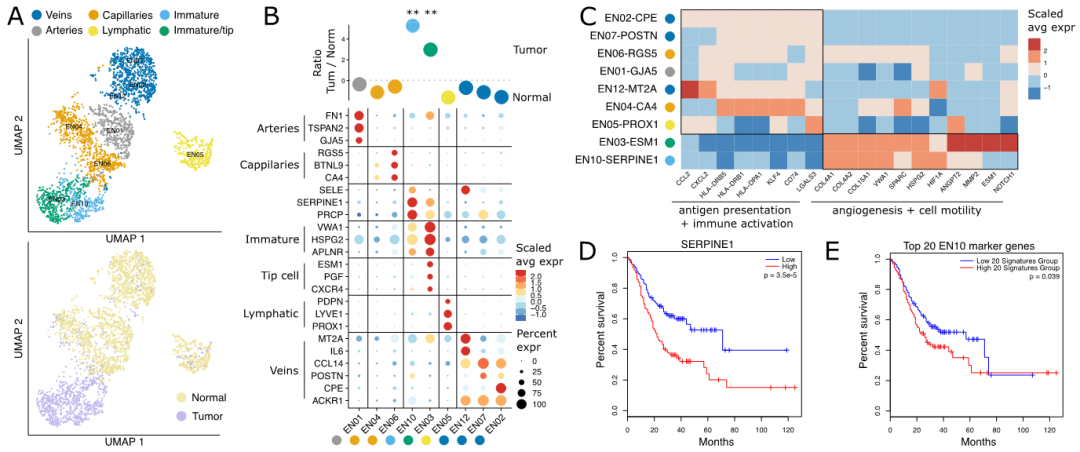
图3 胃癌中混合的内皮细胞状态

图4 促炎和抗炎巨噬细胞呈负相关
肿瘤和正常样本中CD8+ T细胞的比例几乎保持不变,相反的是胃肿瘤样本中CD4+ T细胞增加了5倍以上。对12905个CD4+ T细胞重聚类分成了8个亚簇,并能鉴定出6种T辅助细胞亚型(图5A)。而CD8+ T细胞和NK细胞可以分成13个亚簇,其中12个对应到8种CD8+ T亚型(图5B)。这些细胞亚群中,免疫抑制的Th17和Treg细胞是肿瘤中增加最多的CD4+ T细胞,而初始CD8+ T细胞减少(图5C),可能反映了T细胞在肿瘤中的激活和扩增。Bulk和单细胞数据整合分析发现一个基因簇U4在胃癌中上调,其偏好性地表达在T01-ICOS中(图5D、E)。
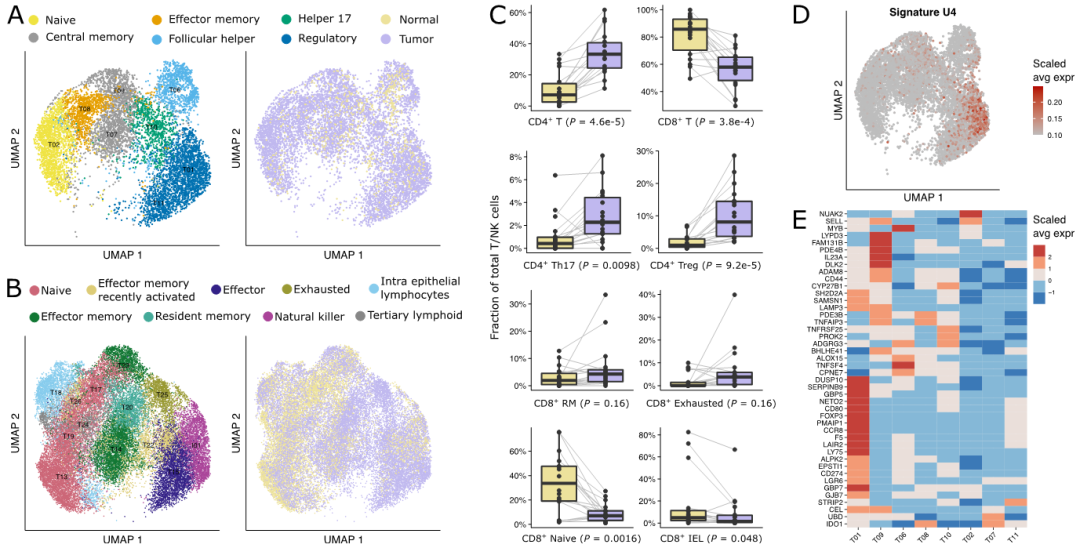
图5 胃癌中免疫抑制性T细胞动态
通过分析配体和受体表达信息建立起胃癌细胞通讯网络,发现F13-CTHRC1在TME网络中表现出最高的中心性指数,与许多细胞亚型有显著的联系(图6A),包括其它的成纤维细胞、脉管相关内皮细胞和免疫细胞(图6B-E)。这些发现表明F13-CTHRC1通过与内皮细胞和免疫细胞的血管生成刺激和免疫抑制相互作用,在胃癌进展中发挥潜在作用。
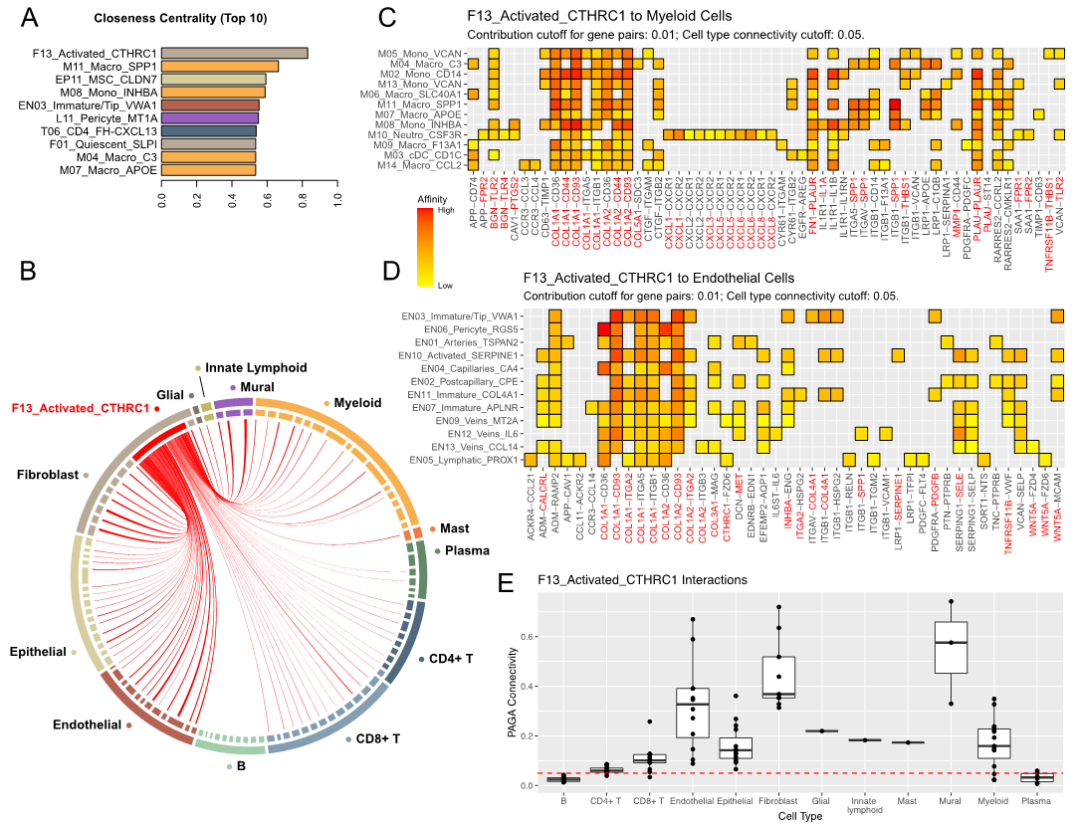
图6 从胃癌单细胞数据推断的细胞通讯显示F13-CTHRC1成纤维细胞的核心作用
作者随后利用单细胞数据作为参考,使用CibersortX推断TCGA样本中每种细胞亚型的比例,随后评估每种细胞亚型高分数和低分数患者之间的生存差异。值得注意的是,肿瘤相关细胞亚型F13-CTHRC1和EN10-SERPINE1比例较高的患者生存时间显著缩短(图7A、B)。相比之下,cDC1树突状细胞的比例(M16-cDC-CLEC9A)对患者的生存有显著的积极影响(图7C)。细胞通讯发现M16-CLEC9A通过一系列通讯信号(最突出的包括XCL1/ XCL2-XCR1受体-配体轴)与广泛的CD8+ T细胞和NK细胞群相互作用(图7D)。因此,结合这些负相关的特征,在F13富集/M16缺失患者和F13缺失/ M16富集患者之间存在更明显的生存差异(图7E)。
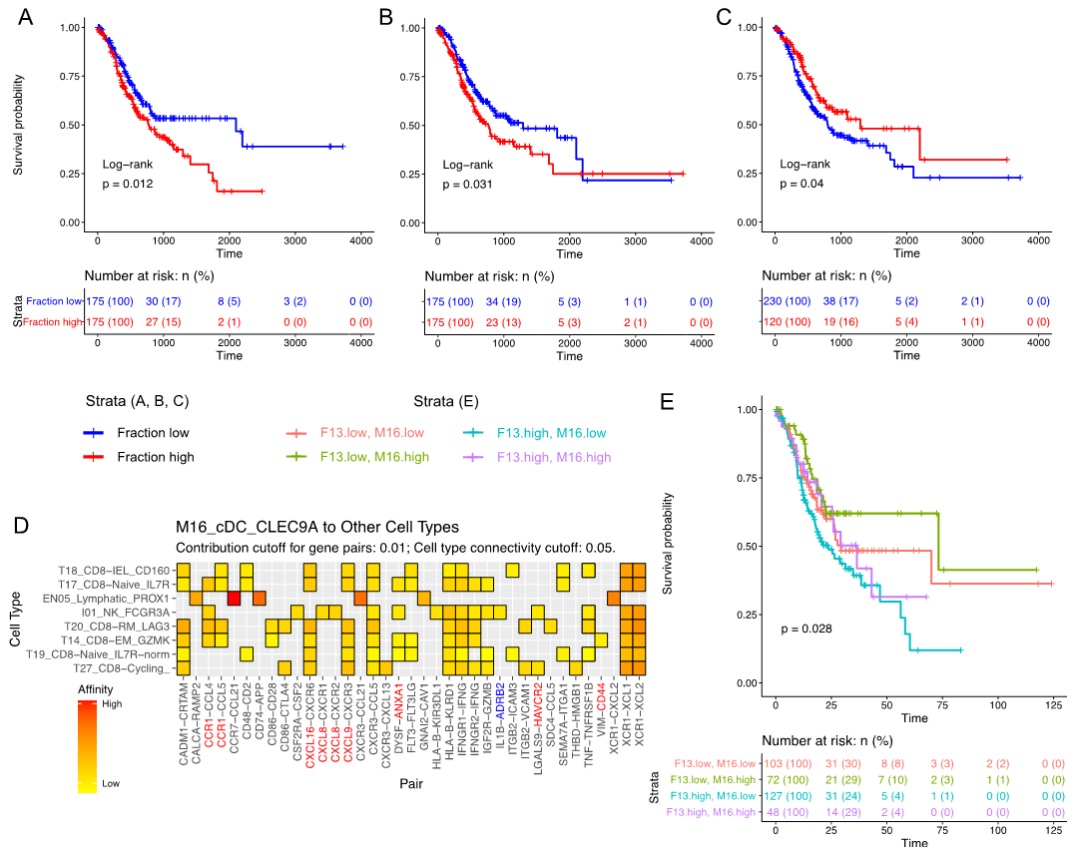
图7 独立胃癌队列的bulk RNA-seq去卷积分析揭示细胞亚型对预后的影响
为了研究在胃TME中确定的11种主要细胞类型或81种亚型中是否有任何一种可以用于预测免疫治疗的反应,作者分析了由每种细胞类型的主要标记基因组成的二元分类模型在上述胃癌免疫治疗队列中预测治疗结果的能力。结果显示在该队列中,T27循环CD8+ T细胞的存在(ROC曲线下面积为0.85)及随后的泛CD8+ T细胞(0.81)的预测治疗结果最佳(图8E)。而F14-ADAM28激活成纤维细胞的存在以及泛成纤维细胞的存在是对无响应的最佳预测,AUROC分别为0.82和0.7(图8E),特异性高达100%。
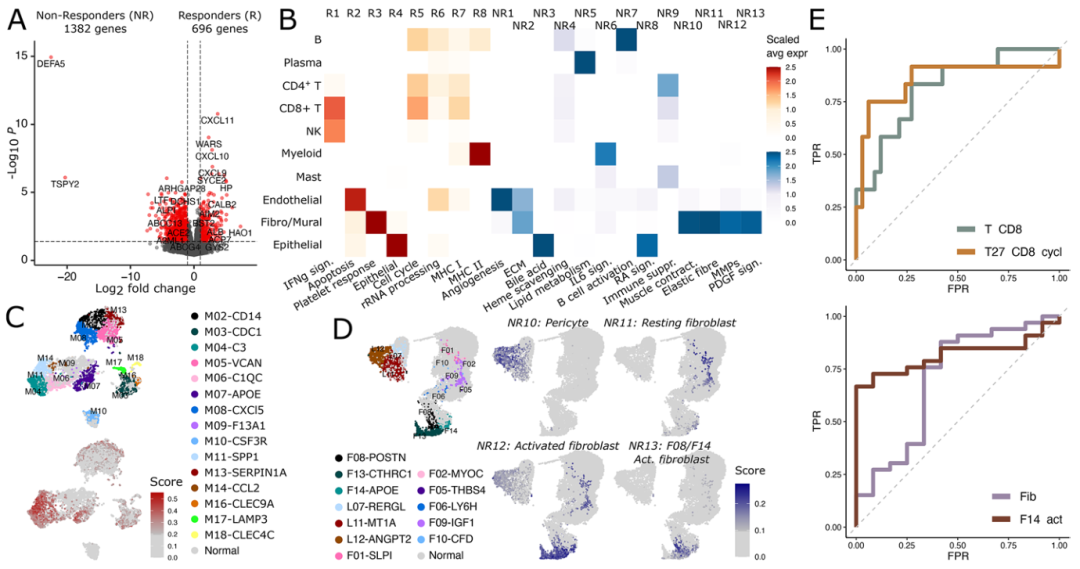
图8 肿瘤免疫治疗应答和非应答基因的细胞起源

