
 咨询热线:400-065-6886
咨询热线:400-065-6886

 咨询热线:400-065-6886
咨询热线:400-065-6886
主题概要
01
加载pheatmap包和数据准备
1. 加载pheatmap包,若未安装,先安装该包
install.packages('pheatmap')
In [1]:
library(pheatmap)
2. 查看数据
df数据,包含3个部分(图形化展示需要用到):
1. 样品名(表头)
2. 基因名(A列)
3. 表达量数值
In [2]:
df = read.table('Differential_Expression_Genes.txt',header = T,row.names = 1,check.names = F)
head(df)
Out[2]:
Ctrl-1 Ctrl-2 Ctrl-3 Case1 Case2 Case3
Ltbp2 13475.1224 10028.49763 11640.97698 28778.9332 28157.054570 28998.069090
Cxcr5 0.0000 0.00000 0.00000 19.8369 17.723921 8.460277
Wnt1 0.0000 0.00000 0.00000 2.2041 8.340669 4.700154
Id2 1598.1884 1660.98059 1233.68103 3408.2732 3857.559222 3188.584361
Id1 1241.1028 1320.58772 1363.78675 2605.2462 3215.327740 2606.705316
1700012B09Rik 126.8728 97.90564 33.68809 362.9418 523.376954 342.171199
3. 使用未归一化数据绘图
In [3]:
pheatmap(df)
Out[3]:
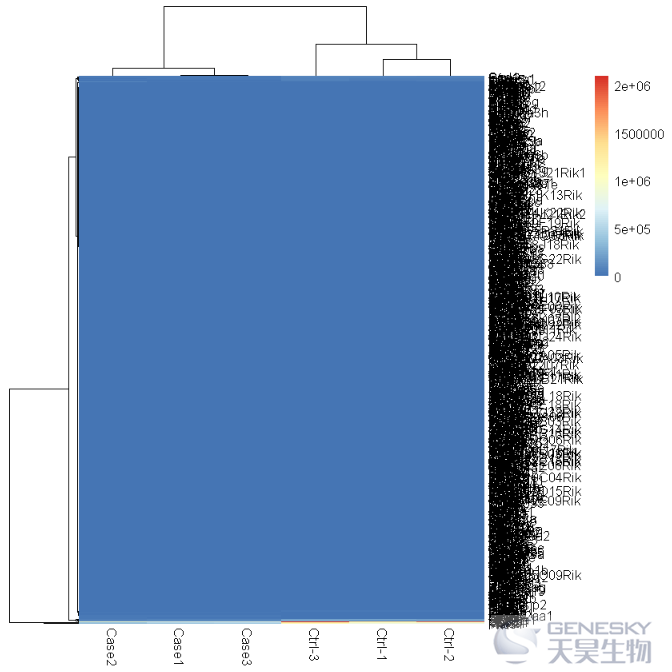
太丑了,不忍直视,我们改进一下吧!!
02
表达量数值处理与展示
1. 矩阵进行标准化(标准化参数为scale,可选"none", "row", "column")
In [4]:
pheatmap(df,scale = 'row')
Out[4]:
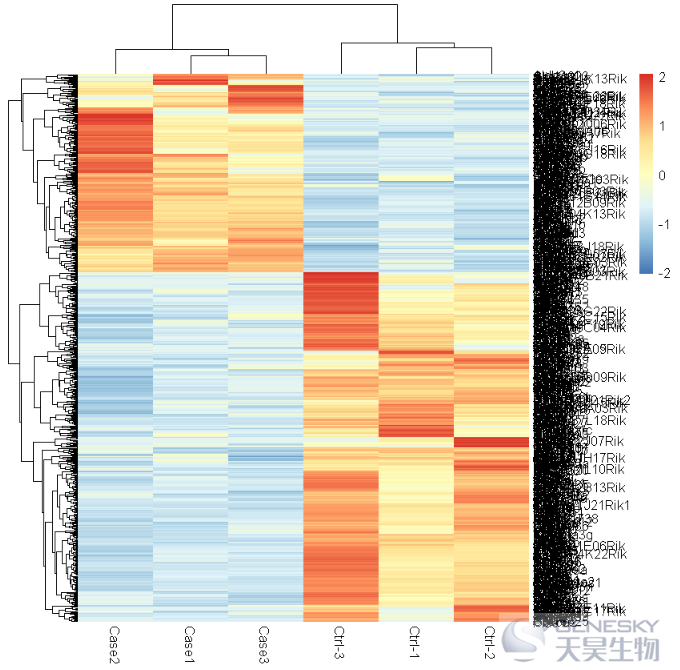
2.更换颜色
In [5]:
pheatmap(df,scale = 'row',color = colorRampPalette(c("green", "black","red"))(100))
Out[5]:
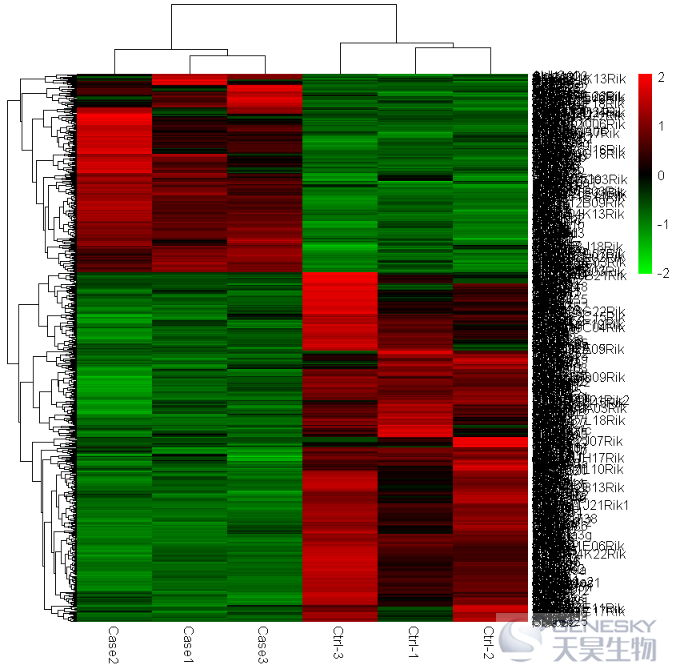
3. 显示数值
为了让数值能看到,这里只选择前20个基因绘图。且颜色使用默认的颜色。
In [6]:
df_20=head(df,20)
In [7]:
df_20
Out[7]:
Ctrl-1 Ctrl-2 Ctrl-3 Case1 Case2 Case3
Ltbp2 13475.122360 10028.497630 11640.976980 28778.9332 28157.054570 28998.069090
Cxcr5 0.000000 0.000000 0.000000 19.8369 17.723921 8.460277
Wnt1 0.000000 0.000000 0.000000 2.2041 8.340669 4.700154
Id2 1598.188392 1660.980593 1233.681025 3408.2732 3857.559222 3188.584361
Id1 1241.102765 1320.587723 1363.786745 2605.2462 3215.327740 2606.705316
1700012B09Rik 126.872830 97.905642 33.688088 362.9418 523.376954 342.171199
Dock8 230.212796 206.057222 216.068428 531.1881 835.109442 623.240398
Cngb3 0.000000 0.000000 0.000000 2.9388 9.383252 5.640185
Srcin1 0.000000 0.000000 0.000000 8.0817 27.107173 0.000000
Pdk4 2825.989973 2500.009173 2288.466685 5480.8619 7235.530000 5462.518785
Gja3 0.000000 0.000000 0.000000 6.6123 15.638754 4.700154
Hmga1 276.255356 225.410663 132.429037 432.7383 613.039141 558.378275
Blnk 475.773113 347.223496 334.557566 1029.3147 689.147742 1227.680181
Ahrr 375.502650 340.392870 363.599021 657.5565 1023.817069 1073.515136
Myocd 0.000000 0.000000 0.000000 2.9388 13.553586 23.500769、
Slco4a1 4.092672 10.245939 4.646633 31.5921 33.362674 29.140954
Hmga2-ps1 475.773113 341.531308 347.335806 809.6394 760.043425 819.706829
Btbd11 1114.229935 628.417606 631.942069 1827.1989 1917.311192 2000.385472
Gm4425 0.000000 0.000000 0.000000 2.2041 4.170334 10.340338
Sult1a1 12.278016 3.415313 15.101557 19.1022 42.745927 39.481292
In [8]:
pheatmap(df_20,scale = 'row',display_numbers = TRUE)
Out[8]:
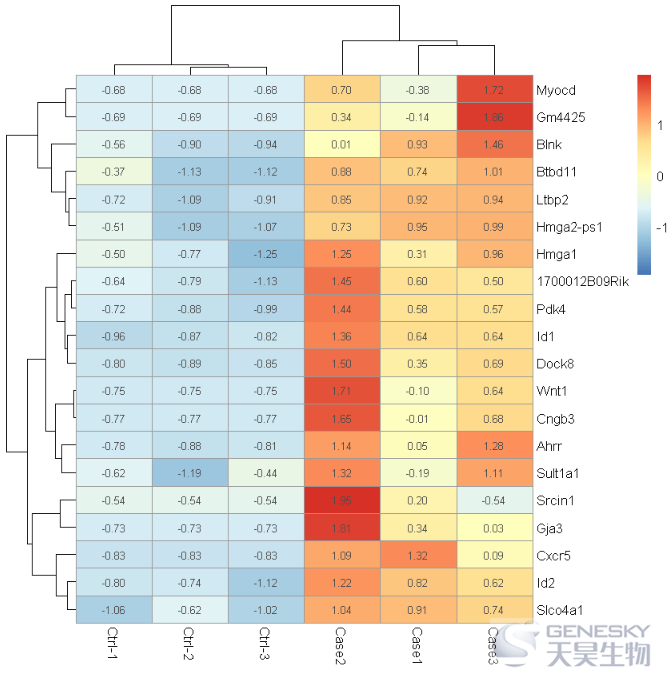
保留1位小数点
In [9]:
pheatmap(df_20,scale = 'row',display_numbers = TRUE,number_format = "%.1f")
Out[9]:
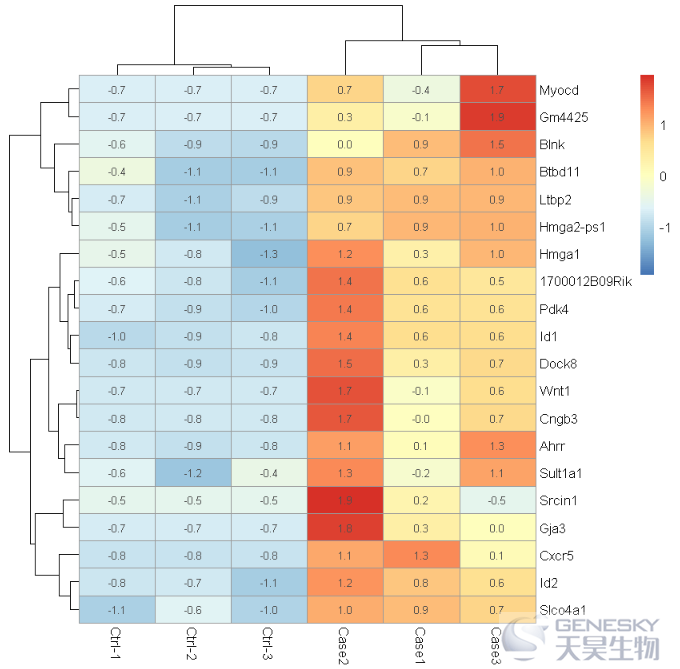
In [10]:
pheatmap(df_20,scale = 'row', display_numbers = matrix(ifelse(df_20 > 0.5, "*", ""), nrow(df_20)))#还可以自己设定要显示的内容;
Out[10]:
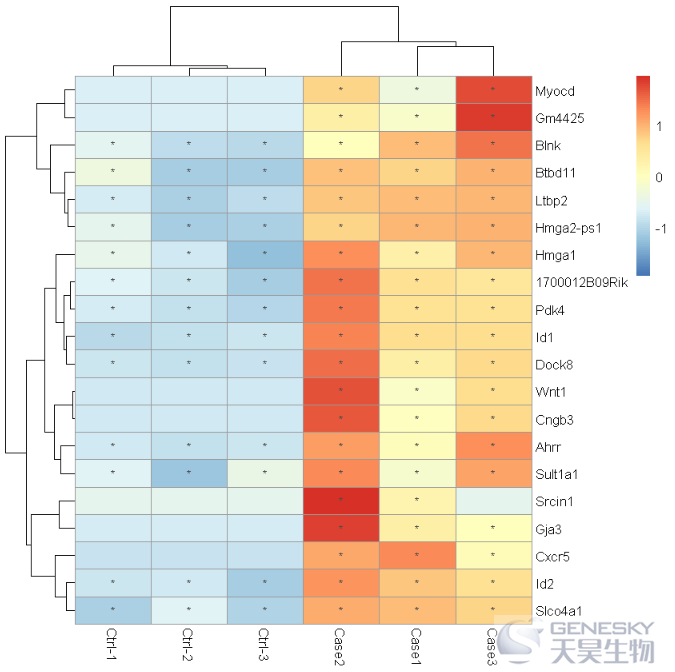
03
样品名和基因名的处理
1. 隐藏样品名或基因名、修改文字显示大小,渐变颜色调整。参数示例如下:
show_rownames = F,
show_colnames=T,
fontsize_row=6, fontsize_col=5
In [11]:
pheatmap(df,scale = 'row',color = colorRampPalette(c("green", "black","red"))(100),
show_rownames = F,show_colnames=T, fontsize_col=20)
Out[11]:
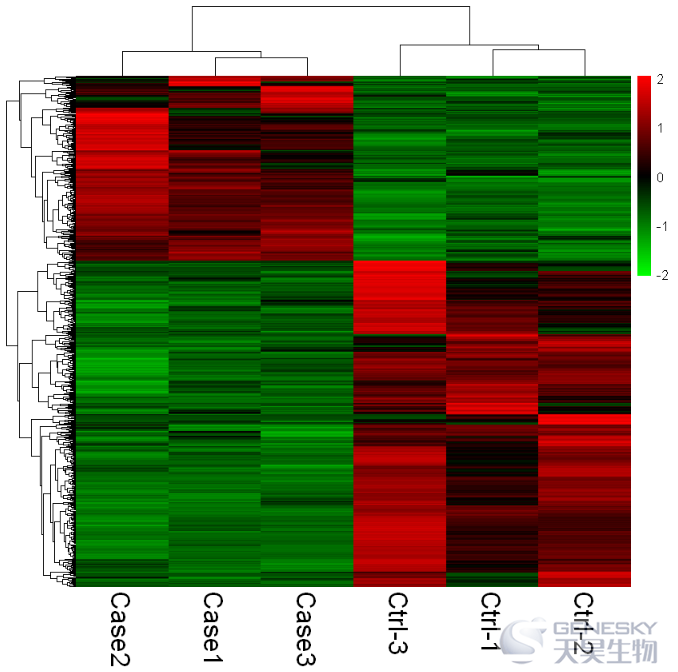
2. 样品或基因不聚类
有时候同一组的样品没有聚在一起,或者基因聚类效果不好看,我们可以选择不聚类,参数如下:
cluster_col =F,cluster_rows = F
In [12]:
pheatmap(df,scale = 'row',color = colorRampPalette(c("green", "black","red"))(100),
show_rownames = F,show_colnames=T, fontsize_col=5,
cluster_col =F,cluster_rows = F)
Out[12]:
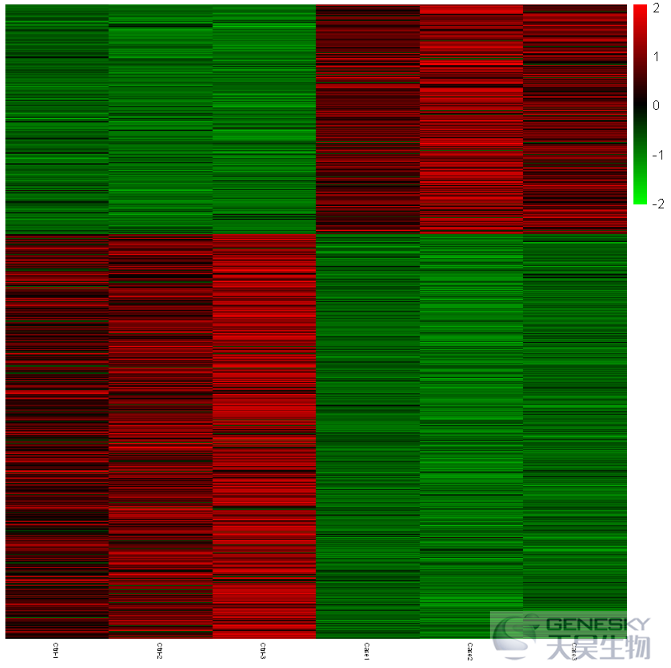
3. 添加样品或基因的分组信息
In [13]:
annotation_col = read.table('sample_group.txt',header = T,row.names = 1)
annotation_col
Out[13]:
group sub_group
Ctrl 1
Ctrl 2
Ctrl 3
Case1 Case 1
Case2 Case 2
Case3 Case 3
In [14]:
annotation_row = read.table('gene_group.txt',header = T,row.names = 1)
head(annotation_row)
Out[14]:
type
Ltbp2 Up
Cxcr5 Up
Wnt1 Up
Id2 Up
Id1 Up
1700012B09Rik Up
In [15]:
pheatmap(df,scale = 'row',color = colorRampPalette(c("green", "black","red"))(100), show_rownames = F,
annotation_col = annotation_col, annotation_row = annotation_row)
Out[15]:
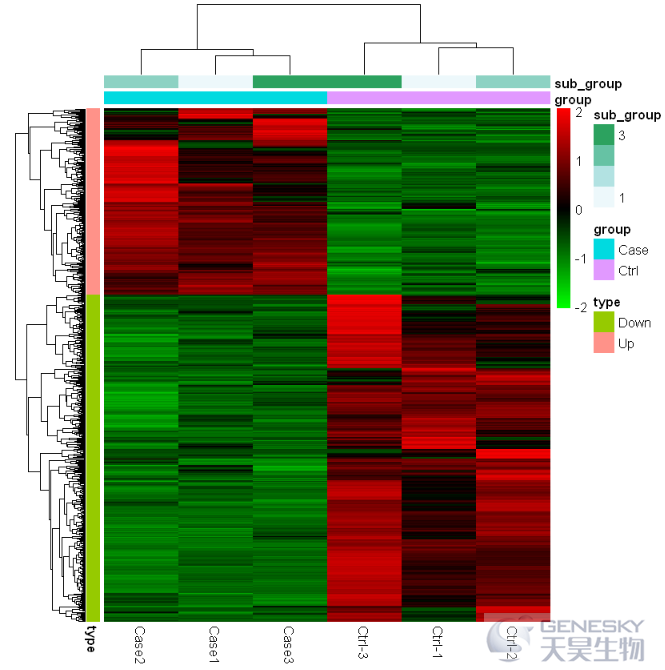
4. 根据行列的聚类数将热图分隔开;
cutree_rows=2,cutree_cols=2 ,根据聚类结果分开;
cluster_rows = FALSE,gaps_row=c(100,500) 指定分开的行,需要行不聚类;
cluster_col =FALSE,gaps_col=c(2,4) 指定分开的列,需要列不聚类。
In [16]:
pheatmap(df,scale = 'row',color = colorRampPalette(c("green", "black","red"))(100), show_rownames = F,
annotation_col = annotation_col, annotation_row = annotation_row,
cutree_rows=2,cutree_cols=2)
Out[16]:
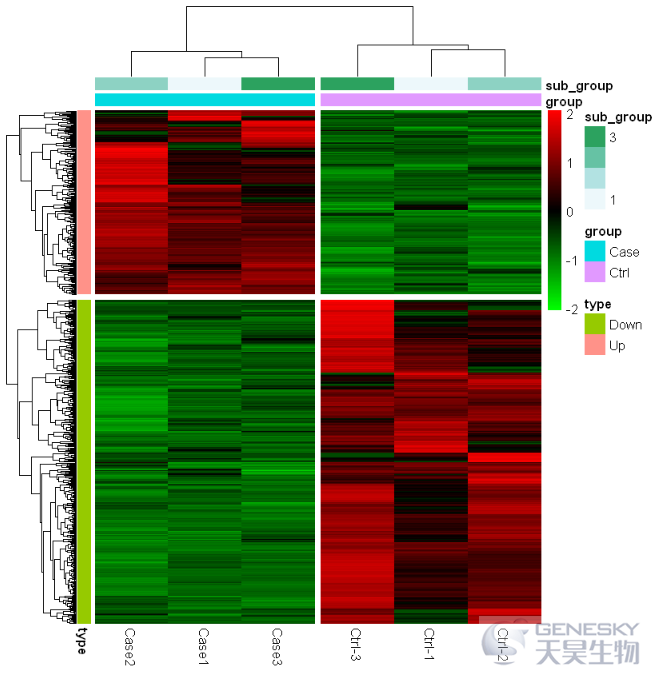
In [17]:
pheatmap(df,scale = 'row',color = colorRampPalette(c("green", "black","red"))(100), show_rownames = F,
annotation_col = annotation_col, annotation_row = annotation_row,
cluster_rows = FALSE,gaps_row=c(100,200))
Out[17]:
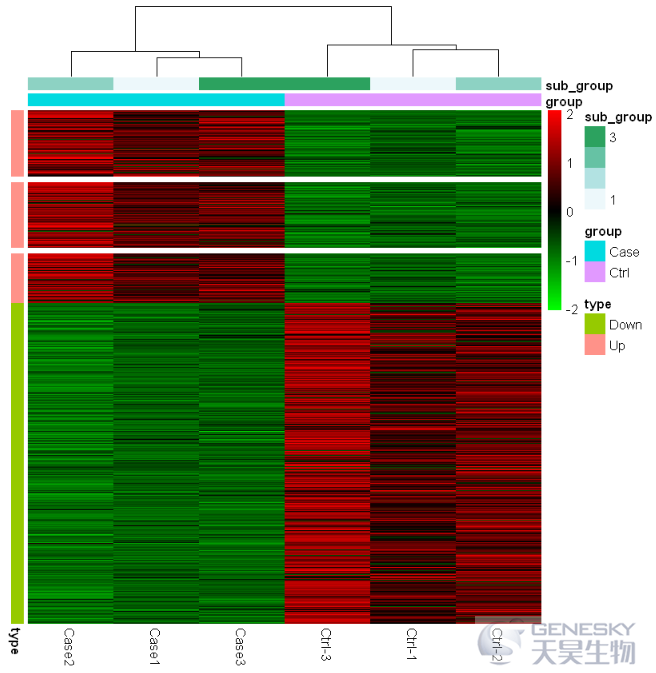
In [18]:
pheatmap(df,scale = 'row',color = colorRampPalette(c("green", "black","red"))(100), show_rownames = F,
annotation_col = annotation_col, annotation_row = annotation_row,
cluster_rows = FALSE,gaps_row=c(100,200),cluster_col =FALSE,gaps_col=c(2,4))
Out[18]:
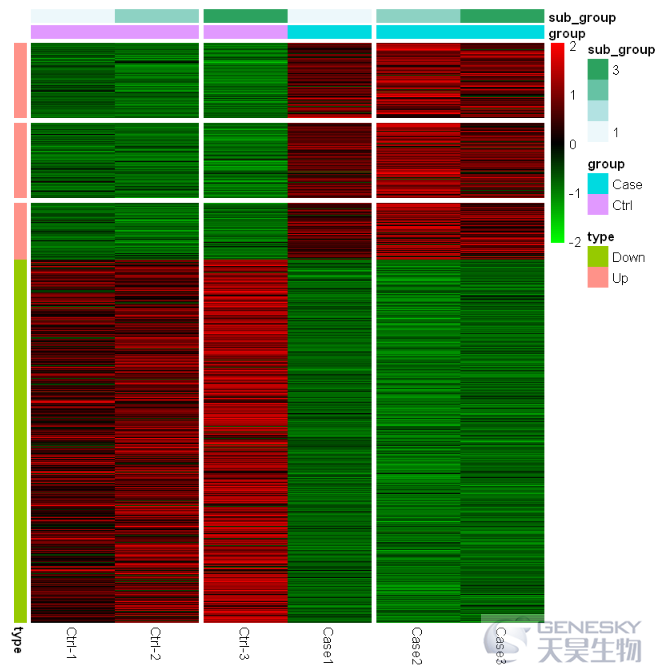
代码和数据下载:
链接:
https://pan.baidu.com/s/1L2B_DDUUvfYzifjgxAb4fg
提取码:mbk2
往期链接:
20分钟搞定GEO上传,史上最简单、最详细的GEO数据上传攻略;
【零基础学绘图】之绘制PCA图(二);
【零基础学绘图】之alpha指数箱体图绘制(一);
如果您对本文案介绍的方法或代码有疑问,
请扫码添加QQ群沟通
【本群将为大家提供】
分享生信分析方案
提供数据素材及分析软件支持
定期开展生信分析线上讲座

QQ号:1040471849
作者:大熊
审核:有才
来源:天昊生信团

创新基因科技,成就科学梦想
微信扫一扫
关注该公众号
前往“发现”-“看一看”浏览“朋友在看”